Article
MS 마커를 활용한 지역별 오계 유전자원의 다양성 및 유연관계 분석
노희종1, 김관우1, 이진욱1, 전다연1, 김승창1, 전익수1, 고응규1, 이준헌2, 김성희3, 백준종4, 오동엽5, 한재용6, 이승숙7, 조창연1,†
Genetic Diversity and Relationship of Ogye Population in Korea Using 25 Microsatellite Markers
Hee-Jong Roh1, Kwan-Woo Kim1, Jin-Wook Lee1, Da-Yeon Jeon1, Seung-Chang Kim1, Ik-Soo Jeon1, Yeoung-Gyu Ko1, Jun-Heon Lee2, Sung-Hee Kim3, Jun-Jong Baek4, Dong-Yep Oh5, Jae-Yong Han6, Seung-Sook Lee7, Chang-Yeon Cho1,†
Author Information & Copyright ▼
1농촌진흥청 국립축산과학원 가축유전자원센터 농업연구관/농업연구사
1Senior Researcher/Researcher, Animal Genetic Resources Center, National Institute of Animal Science, RDA, Namwon 55717, Republic of Korea
2Professor, Division of Animal and Dairy Science, Chungnam National University, Daejeon 34134, Republic of Korea
3Officer, Chungbuk Institute of Livestock and Veterinary Research, Cheongju 28153, Republic of Korea
4Researcher, Chungnam Institute of Livestock Experiment Research, Cheongyang 33350, Republic of Korea
5Researcher, Gyeongbuk Livestock Research Institute, Yeongju 36052, Republic of Korea,
6Professor, Department of Agricultural Biotechnology and Research Institute of Agriculture and Life Sciences, Seoul National University, Seoul 08826, Republic of Korea
7Representative, Jisan Farm, Nonsan 29009, Republic of Korea
ⓒ Copyright 2018, Korean Society of Poultry Science. This is an Open-Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Aug 13, 2018 ; Revised: Sep 13, 2018 ; Accepted: Sep 13, 2018
Published Online: Sep 30, 2018
적요
본 연구는 연산오계(천연기념물 제265호)와 이를 기원으로 하는 5개 지역별 오계 집단의 유전적 특성 및 차별성을 분석하기 위해 25개의 초위성체(MS) 마커를 이용하여 총 9개 집단 243수를 대상으로 유전자형을 분석하였다. 마커별 다형성 분석 결과, 총 153개의 대립유전자가 확인되었으며, Hexp와 PIC의 경우 MCW0145에서 각각 0.640, 0.570으로 가장 높았고, Hobs는 MCW0252에서 0.607로 가장 높은 값을 나타내었다. 반면, LEI0166에서 Hexp, Hobs, PIC가 각각 0.248, 0.204, 0.202로 가장 낮았다. 집단간 유전거리 분석 결과로는 9개 집단중 YSO 집단과 SUO 집단이 가장 가까운(0.073) 반면, LG 집단과 CBO 집단 사이에서 가장 먼(0.937) 것으로 확인되었다. 집단의 실제 구조를 확인하기 위한 집단별 균일도를 분석한 결과, 공시된 9개의 집단은 3개의 집단으로 구분했을 때 최적의 K값(7.96)을 얻을 수 있었으며, 5개의 오계 집단(YSO, ARO, CBO, CNO, SUO) 및 LG 집단과 CN․RIR 집단은 각각 1, 2, 3번 군집에 분포하고 있는 것으로 나타났다. 한편, GBO 집단의 경우 1번과 3번 클러스터에 걸쳐서 분포하고 있는 것으로 보아 사육과정에서 타집단과의 교잡이 일어났을 것으로 추정된다. 이러한 결과를 통해 추후 오계 유전자원에 대한 국가 수준의 유전적 특성평가 및 관리의 기초 자료로 유용하게 활용될 것으로 기대된다.
ABSTRACT
The aim of this study was to evaluate the genetic diversity and relationships of Ogye populations in Korea. A total of 243 genomic DNA samples from 6 Ogye population (Yeonsan Ogye; YSO, Animal Genetic Resources Research Center Ogye; ARO, Chungbuk Ogye; CBO, Chungnam Ogye; CNO, Gyeongbuk Ogye; GBO, Seoul National University Ogye; SUO) and 3 introduced chicken breeds (Rhode Island Red; RIR, White Leghorn; LG, Cornish; CN) were used. Sizes of 25 microsatellite markers were decided using GeneMapper Software(v 5.0) after analyzing ABI 3130XL. A total of 153 alleles were observed and the range was 2 to 10 per each locus. The mean of expected and observed heterozygosity and PIC (Polymorphism Information Content) value was 0.53, 0.50, 0.46 respectively. The lowest genetic distance (0.073) was observed between YSO and SUO, and the highest distance (0.937) between the RIR and CBO. The results of clustering analysis suggested 3 clusters (ΔK=7.96). Excluding GBO population, 5 Ogye populations (YSO, ARO, CBO, CNO, SUO) were grouped in same cluster with high genetic uniformity (0.990, 0.979, 0.989, 0.994, 0.985 respectively). But GBO population was grouped in cluster 1 with low genetic uniformity (0.340). The results of this study can be use to basic data for the genetic evaluation and management of Ogye populations in Korea.
Keywords: chicken; Ogye; genetic diversity; polymorpism; microsatellite
서 론
오계의 오(烏)자는 한자로 ‘까마귀’를 뜻하며, 깃털, 피부, 뼈 등이 모두 까마귀처럼 검어서 붙여진 이름으로, 흑계와는 구분되는 품종이다(Lee, 2010). 오계의 기원 역시 다른 닭들과 마찬가지로 동남아시아 지역에서 비롯된 것으로 추정된다. 하지만, 언제 어떻게 도입되었는지에 대해서는 확실하게 알려진 바가 없으나, 이시진의 본초강목(本草綱目), 허준의 동의보감(東醫寶鑑) 등에 그 약효와 쓰임새가 기록되어 있는 것으로 보아, 선조대 이전부터 사육되기 시작한 것으로 추정된다. 현재 국내에 존재하는 대부분의 오계의 경우 혼합종이지만, 천연기념물 제265호(1980년 지정)이며, 세계식량농업기구(FAO) 가축다양성시스템(DAD-IS)에 등재되어 있는 충남 논산시 연산 화악리 지역의 오계(연산오계)는 순수품종으로 보존되고 있다. 이러한 연산오계의 보존을 위해서 국립축산과학원에서는 가축유전자원 국가 보존체계의 확립의 일환으로 지자체 축산연구기관 및 대학을 포함하여 총 12개의 가축생명관리기관을 지정하여 운영하고 있으며, 이 중 4개 기관(충북, 충남, 경북, 서울대)에서는 2000년대 이후 연산오계를 도입하여 지속적으로 세대를 갱신하며 보존해오고 있다. 하지만, 각 지역에 흩어져 세대갱신이 지속된다면 같은 품종임에도 불구하고, 해당지역의 환경조건에 맞게 적응함으로써 초기 집단과는 유전적으로 차이를 보일 것으로 추정된다. 또한, 각 지역에 분포되어 있는 오계 집단이 순수한 집단으로서 적절하게 유지되고 있는지를 살펴볼 필요성이 있다.
최근 품종 및 개체간 유전적 특성을 구명하기 위하여 DNA marker를 활용하는 여러 방법이 제시되고 있다. 그 중 Microsatellite(MS) marker는 2∼6개의 짧고 단순한 염기서열이 반복되는 구조로, 각 품종 및 개체간 염기서열의 반복수에서 차이가 나기 때문에 유전적 다양성이 높다고 알려져 있으며, 유전자 전체에 빈번하게 존재하기에 친자확인이나 품종 및 개체간 유전적 다양성 및 유연관계 등의 분석에 널리 이용되고 있다(Barker et al., 1997; Peelman et al., 1998; Martin et al., 1999; Li et al., 2000; Bjornstad et al., 2003). 현재까지 오계를 포함하여 우리나라의 재래닭 및 외래토착종을 대상으로 MS 마커를 이용한 연구가 다수 보고되어 왔으며, 오계 집단은 재래닭 및 외래토착종 집단과는 다른 하나의 순수한 집단으로 뚜렷하게 구분되는 것으로 나타났다(Lee et al., 2010; Suh et al., 2014; Seo et al., 2015). 하지만 각 지역에 분포되어 있는 오계 집단간의 유전적 특징을 분석한 연구는 전무한 실정이다. 따라서 본 연구에서는 연산오계 집단과 각 지역별로 분포되어 보존되고 있는 5개 집단 오계 유전자원의 유전적 다양성 및 유연관계를 분석하기 위해 25개의 MS 마커를 이용해 집단별 유전자형을 분석하였고, 이를 토대로 각 지역의 오계 집단의 유전적 차이점을 구명해 봄으로써 초기 집단과는 어떤 차이점을 나타내는지를 분석하고자 실시하였다.
재료 및 방법
1. 공시재료 및 MS 마커
본 연구에 이용된 공시재료로는 연산오계 집단과 국립축산과학원 가축유전자원센터 및 각 지역의 4개 기관(충북, 충남, 경북, 서울대)에서 보존되고 있는 오계 집단과 대조구로 외래 도입 토착종 3개 집단(레그혼, 코니시, 로드아일랜드), 총 9개 집단 243수를 이용하였으며(Table 1), 익하정맥으로부터 채혈한 혈액을 이용하여 Genomic DNA Purification Kit (Promega, USA)를 통해 DNA를 추출하였다.
Table 1.
Description of the 9 chicken breeds used in this study
| Abbr. |
Breed |
No. of sample |
Genetic orgin |
Source |
| YSO |
Ogye |
27 |
Korea |
Jisan Farm |
| ARO |
Ogye |
27 |
Korea |
NIAS
|
| CBO |
Ogye |
27 |
Korea |
CILVR |
| CNO |
Ogye |
27 |
Korea |
CILER |
| GBO |
Ogye |
27 |
Korea |
GLRI |
| SUO |
Ogye |
27 |
Korea |
SNU |
| RIR |
Rhode Island Red |
27 |
USA |
NIAS |
| LG |
White Leghorn |
27 |
Italy |
NIAS |
| CN |
Cornish |
27 |
UK |
NIAS |
Download Excel Table
집단별 다양성 분석을 위해 사용한 MS marker는 ISAG/ FAO(FAO, 2004)에서 권고하고 있는 MS marker 10종(ADL 0278, LEI0094, LEI0166, LEI0192, MCW0016, MCW0037, MCW0078, MCW0206, MCW0295, MCW0330)과 Roslin Bioinfomatics Group에서 제공하는 ArkDB database website를 참고하여 다양성이 높게 보고된 15종(ADL0176, ADL0262, ADL0267, LEI0092, LEI0096, LEI0099, LEI0135, MCW0103, MCW0145, MCW0193, MCW0214, MCW0233, MCW0240, MCW0252, MCW0301)의 마커를 선별하여 총 25종을 이용 하였다.
2. PCR 및 유전자형 분석
추출된 DNA는 UV Spectrophotometer(NA-1000, Nanodrop)을 이용해 농도를 측정한 후, 최종 10 μL/μg이 되도록 증류수에 희석하여 PCR 증폭을 위한 주형으로 사용하였다. 각 마커별 증폭산물의 크기와 형광표지 등을 고려하여 2∼6개씩 Multiplex 조합을 만든 후, DNA 중합효소, dNTP, KCL, MgCl2 등이 포함되어 있는 AccuPowerTM Negative dye PCR PreMix(Bioneer, Korea)를 이용하여 주형 DNA 2ul, primer는 각각의 multiplex 조합별로 조건에 맞게 0.4∼2.6 μL(2 pmole/μL)를 첨가하고, 최종부피가 20 μL가 되도록 증류수를 첨가한 후, multiplex PCR을 수행하였다. PCR 반응은 GeneAmp PCR system 9700(Applied Biosystems, USA)을 이용하여 수행하였으며, 95℃에서 5분간 pre-denaturation, 95℃에서 60초간 denaturation, 각 조합에 따라 55∼62℃에서 45초간 annealing, 72℃에서 60초간 extension을 35회 반복한 후 72℃에서 30분 동안 final extension을 실시하였다.
증폭된 PCR 산물은 Hi-Di Foramide(Applied Biosystems, USA)와 GeneScan 500Liz Size Standard(Applied Biosystems, USA)를 혼합하여 10분 동안 95℃에서 변성(denaturation)시킨 후 즉시 얼음에 넣어 상태를 유지하게 하였다. 그 다음 자동 염기 서열 분석 장치인 ABI PRISM 3130XL Genetic Analyzer (Applied Biosystems, USA)에 장착하여 POP 7 polymer (Applied Biosystems, USA)상에서 모세관 전기영동을 통해 분석하였다. 각 마커별 대립유전자들의 크기는 GeneMapper version 5.0 software(Applied Biosystems, USA)를 이용하여 결정하였고, 크기가 결정된 좌위들은 공시된 개체별로 Microsoft Excel(Microsoft, ver 2010, USA)을 이용하여 자료를 취합한 후 각 통계분석 방법에 적용하였다.
3. 통계분석
크기가 결정된 마커별 대립유전자들은 MS tool Kit(Park, 2000)을 이용하여 마커 및 집단별 관측이형접합도(Hobs), 기대이형접합도(Hexp), 다형정보지수(PIC) 등을 계산하였다. 집단간 유전 거리 추정을 위해서는 PHYLIP program pakage (Felesenstein, 2010)을 통해 Nei 등(1983)의 방법을 이용하여 산출하였으며, 이를 토대로 집단별 Neighbor-Joining phylogenetic tree를 작성하였다.
집단의 유전적 균일도 추정은 STRUCTURE software (Pritchard et al, 2000)을 이용하였으며, 구분 가능한 집단 수(K값)을 추정하기 위해서 K를 2부터 9까지 설정하고, 각 값에 대한 평균 추정치 Pr(X│K)와 표준편차를 계산하고, burn-in 20,000회, MCMC (markov chain monte carlo) 반복수 100,000회, 이를 10회 반복하여 계산된 결과를 Evanno 등(2005)의 방법이 적용된 STRUCTURE HARVESTER(Earl and von-Holdt, 2012)를 사용하여 ΔK 값을 계산하여 최적의 K값을 결정하였으며, CLUMMP(Jakobsson and Rosengerg, 2007)를 이용해 10회 반복하여 나온 최적의 K값에 대한 cluster별 유전적 균일도를 계산하였고, 이후 각 cluster에 대한 집단의 실제 분포를 DISTRUCT(Rosenberg, 2004)를 이용하여 도식화하였다.
결과 및 고찰
1. 마커 및 집단별 유전적 다양성
25개 MS 마커를 이용하여 6개 오계 집단 및 3개 외래토착종 집단을 포함하여 총 9개 집단 243수에 대한 마커별 대립유전자 수(NA), 기대이형접합도(Hexp), 관측이형접합도(Hobs), 다형정보지수(PIC)를 Table 2에 제시하였다. 25개의 마커에서 2∼10개의 범위로 총 153개의 대립유전자가 나타났으며, 마커별 평균 6.12개의 대립유전자를 보유하고 있는 것으로 확인되었다. MCW0145의 경우, Hexp와 PIC가 각각 0.640, 0.570으로 가장 높게 나타났으며, Hobs는 MCW0240에서 0.638로 가장 높게 나타났다. 반면, LEI0166에서는 Hexp, Hobs, PIC 각각 0.248, 0.204, 0.202로 가장 낮은 수치를 보이고 있었다. 한편, MCW0145의 경우, 우리나라 재래닭 및 외래토착종을 대상으로 진행한 여러 논문(Kong et al., 2006; Seo et al., 2013; Suh et al., 2014)에서도 높은 다형성을 나타내고 있는 것으로 보고되었다.
Table 2.
The statistical analysis of heterozygosity, polymorphism information content using 25 MS markers
| Locus |
NA |
Hexp |
Hobs |
PIC |
| ADL0176 |
7 |
0.603 |
0.523 |
0.523 |
| ADL0262 |
3 |
0.444 |
0.425 |
0.376 |
| ADL0267 |
8 |
0.593 |
0.453 |
0.524 |
| ADL0278 |
5 |
0.542 |
0.527 |
0.471 |
| LEI0092 |
7 |
0.636 |
0.626 |
0.569 |
| LEI0094 |
7 |
0.567 |
0.578 |
0.500 |
| LEI0096 |
8 |
0.520 |
0.446 |
0.469 |
| LEI0099 |
5 |
0.535 |
0.466 |
0.464 |
| LEI0135 |
5 |
0.391 |
0.285 |
0.309 |
| LEI0166 |
4 |
0.220 |
0.181 |
0.180 |
| LEI0192 |
10 |
0.633 |
0.612 |
0.570 |
| MCW0016 |
5 |
0.471 |
0.484 |
0.398 |
| MCW0037 |
4 |
0.556 |
0.547 |
0.482 |
| MCW0078 |
2 |
0.276 |
0.284 |
0.223 |
| MCW0103 |
4 |
0.415 |
0.383 |
0.321 |
| MCW0145 |
9 |
0.640 |
0.593 |
0.570 |
| MCW0193 |
9 |
0.601 |
0.566 |
0.545 |
| MCW0206 |
6 |
0.496 |
0.500 |
0.428 |
| MCW0214 |
10 |
0.622 |
0.603 |
0.553 |
| MCW0233 |
4 |
0.564 |
0.584 |
0.486 |
| MCW0240 |
8 |
0.639 |
0.638 |
0.569 |
| MCW0252 |
6 |
0.629 |
0.607 |
0.550 |
| MCW0295 |
5 |
0.528 |
0.525 |
0.457 |
| MCW0301 |
8 |
0.514 |
0.528 |
0.445 |
| MCW0330 |
4 |
0.582 |
0.611 |
0.499 |
Download Excel Table
Table 3에서는 집단별 다양성 분석 결과를 제시하였다. 가장 많은 대립유전자를 보유하고 있는 집단은 YSO(4.08)로 나타났으며, RIR 집단(2.84)에서 가장 적은 대립유전자를 보유하고 있는 것으로 확인되었다. Hexp와 Hobs의 경우, SUO 집단에서 각각 0.607, 0.601로 가장 높은 수치를, LG 집단에서 0.399, 0.361로 가장 낮은 수치를 보이고 있었으며, 전반적으로 6개 오계 집단이 외래토착종에 비해 높은 다양성을 나타내고 있었다. 이는 외래토착종의 경우, 우리나라에 도입된 뒤 수십 년 이상의 세대갱신을 지속함으로써 유전적 고정화 작업이 이루어졌기 때문인 것으로 사료된다.
Table 3.
Genetic diversity parameters in 9 chicken population
| Population |
Sample size |
Marker |
Hexp |
Hobs |
MNA |
| YSO |
27 |
25 |
0.595±0.035 |
0.539±0.019 |
4.08±1.44 |
| ARO |
27 |
25 |
0.537±0.038 |
0.495±0.019 |
3.28±1.06 |
| CBO |
27 |
25 |
0.558±0.033 |
0.582±0.019 |
3.24±1.09 |
| CNO |
27 |
25 |
0.535±0.037 |
0.514±0.019 |
3.68±1.35 |
| GBO |
27 |
25 |
0.562±0.027 |
0.473±0.019 |
3.88±1.27 |
| SUO |
27 |
25 |
0.607±0.035 |
0.601±0.019 |
3.96±1.34 |
| RIR |
27 |
25 |
0.474±0.036 |
0.479±0.019 |
2.84±0.90 |
| LG |
27 |
25 |
0.399±0.047 |
0.361±0.018 |
3.00±1.32 |
| CN |
27 |
25 |
0.491±0.035 |
0.481±0.019 |
3.00±0.82 |
Download Excel Table
2. 집단간 유전거리 및 유연관계
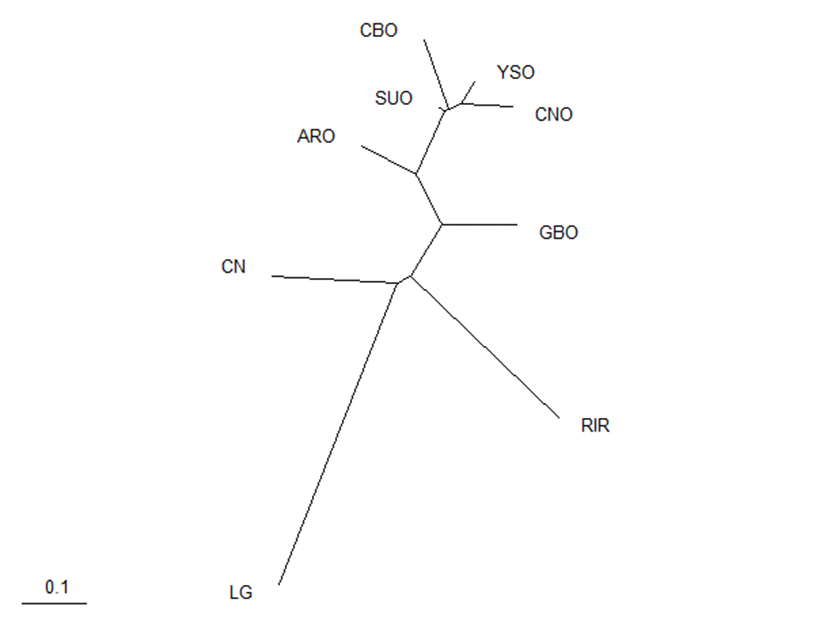
공시된 9개 집단간의 유전적 거리를 추정하기 위해 마커별 대립유전자의 빈도를 바탕으로 PHYLIP program package를 이용하여 Nei 등(1978)의 방법을 통해 계산된 집단간 유전거리를 Table 4에 제시하였다. 분석 결과, YSO 집단과 SUO 집단 사이에서 0.073으로 가장 가까운 것으로 나타났으며, 외래토착종인 LG 집단과 CBO 집단 사이에서 0.937로 가장 먼 유전거리를 보이고 있었다. 특히 오골계 집단의 경우, 서로 0.5이내의 범위의 유전거리를 나타내고 있었으며, 3개의 외래토착종 중 CN 집단과 가장 가까운 유전거리를, LG 집단과 가장 먼 유전거리를 나타내고 있음을 확인하였다. 오재돈 등(2008), 이풍연 등(2011), 서상원 등(2014)의 보고에서도 오계 집단이 외래토착종 중 코니시 집단과 가장 가까운 유전거리를 형성하고 있는 것으로 보고되었다.
Table 4.
Matrix of Nei genetic distances observed among 9 chicken populations
|
|
YSO |
ARO |
CBO |
CNO |
GBO |
SUO |
RIR |
LG |
| ARO |
0.259 |
|
|
|
|
|
|
|
| CBO |
0.175 |
0.300 |
|
|
|
|
|
|
| CNO |
0.114 |
0.316 |
0.209 |
|
|
|
|
|
| GBO |
0.352 |
0.295 |
0.448 |
0.398 |
|
|
|
|
| SUO |
0.073 |
0.208 |
0.128 |
0.101 |
0.320 |
|
|
|
| RIR |
0.661 |
0.630 |
0.679 |
0.757 |
0.501 |
0.603 |
|
|
| LG |
0.822 |
0.813 |
0.937 |
0.928 |
0.846 |
0.770 |
0.811 |
|
| CN |
0.548 |
0.411 |
0.679 |
0.647 |
0.387 |
0.527 |
0.572 |
0.694 |
Download Excel Table
각 집단의 분포를 확인하기 위해 유전적 거리를 바탕으로 PHYLIP program package을 이용하여 집단간 Neighbor-Joining phylogenetic tree를 작성하여 Fig. 1에 제시하였다. 분석 결과, 3개의 외래토착종 집단이 각각 별도의 가지를 형성하고 있었으며, 오계 집단의 경우 집단간 큰 차이를 보이지는 않았지만, GBO 집단의 경우 비교적 별도의 그룹을 형성하고 있음을 확인하였다.
Fig. 1.
Neighbor-Joining Phylogenetic tree showing genetic relationships among 9 chicken populations based on Nei distances.
YSO: Yeonsan Ogye, ARO: Animal Genetic Resources Research Center Ogye, CBO: Chungbuk Ogye, CNO: Chungnam Ogye, GBO: Gyengbuk Ogye, SUO: Seoul National University Ogye, RIR: Rhode Island Red, LG: White Leghorn, CN: Cornish
Download Original Figure
3. 집단의 균일도
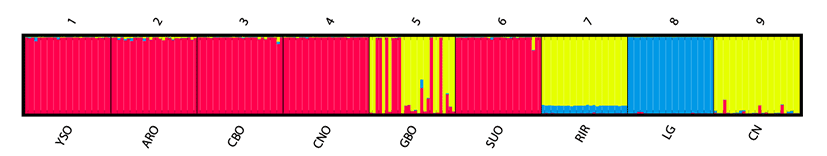
공시된 9개 집단이 실제로 몇 개의 군집으로 구분이 가능한지를 살펴보기 위해 Bayesian clustering법이 적용된 Structure software를 이용해 K값을 2부터 9까지 설정하였고, 분석 후 나온 데이터를 바탕으로 STRUCTURE HARVESTER를 이용해 Evanno(2005)의 방법으로 최적의 K값을 계산한 결과, K=3에서 가장 높은 ΔK값(7.96)을 얻을 수 있었다. Table 5에서는 K=3일 때의 각 집단별 유전적 균일도를 나타내었으며, Fig. 2에서는 집단별 군집 형성 양상을 도식화하여 나타내었다. 분석 결과 YSO, ARO, CBO, CNO, SUO 집단은 1번 군집에서 97% 이상의 매우 높은 균일도를 보였으나, GBO 집단의 경우 34.0%의 균일도를 나타내었고, 3번 군집에서 65.3%로 가장 높은 균일도를 나타내고 있었다. 외래 토착종의 경우, CN 집단이 3번 군집에서 97.3%, LG 집단이 2번 군집에서 99.5%로 높은 균일도를 보이고 있었으며, RIR 집단의 경우 3번 군집에서 89.6%의 균일도를 나타내고 있었다. 이는 Fig. 1에서의 결과와 매우 유사한 양상을 보이는 것으로 확인되었다. 한편, 유전거리 분석 결과에서는 오계집단과 코니시 집단이 비교적 가까운 거리를 나타내고 있었지만, 균일도 분석 결과 서로 뚜렷하게 구분되는 집단임을 확인할 수 있었다. 이상의 결과로 오계집단의 경우, 외래토착종과는 구분 되는 품종임을 확인할 수 있었다. 또한, GBO 집단을 제외한 YSO 집단 및 5개 지역의 오계 집단은 하나의 군집을 형성하고 있었으며, GBO 집단의 경우 두 개(Cluster 1, 3)의 군집에 걸쳐 분포하는 것을 확인할 수 있었다. GBO 집단은 나머지 4개 지역 오계 집단과 같은 연산오계에서 유래된 집단이며, 표현형적 특징에서도 타집단과의 차이점이 나타나지 않았다. 하지만 유전적인 특징에서는 타집단과는 서로 다른 양상을 나타내고 있었으며, 이는 일부 개체에서 타집단과의 교잡이 일어나 균일도가 떨어진 것으로 보인다. 따라서, 추후 균일도를 높이기 위해 개체 식별 및 계획 교배를 통한 지속적인 유전적 고정화 작업이 필요할 것으로 사료된다. 전체적으로, 각 지역별 오랜 시간 분포되어 사육되었기 때문에 유전적 다양성 측면에서는 다소 차이를 보이고 있었으나, GBO 집단을 제외하면 유전적 균일도 측면에서 큰 차이점이 없는 것으로 보아, 각 지역에서 적절하게 계통을 유지하며 사육되었음을 확인할 수 있었으며, 국가 수준의 유전자원 보존을 위해 지속적인 관리가 필요할 것으로 사료된다.
Fig. 2.
Clustering assignment of the 9 chicken populations obtained by STRUCTURE analysis (K=3).
YSO: Yeonsan Ogye, ARO: Animal Genetic Resources Research Center Ogye, CBO: Chungbuk Ogye, CNO: Chungnam Ogye, GBO: Gyengbuk Ogye, SUO: SEoul National University Ogye, RIR: Rhode Island Red, LG: White Leghorn, CN: Cornish.
Download Original Figure
Table 5.
Population of membership of each 9 chicken breed genotypes with 25 microsatellite markers in the 3 inferred clusters using STRUCTURE analysis
| Population |
Inferred cluster |
Number of individual |
| 1 |
2 |
3 |
| YSO |
0.990
|
0.006 |
0.004 |
27 |
| ARO |
0.979
|
0.007 |
0.014 |
27 |
| CBO |
0.989
|
0.004 |
0.008 |
27 |
| CNO |
0.994
|
0.003 |
0.003 |
27 |
| GBO |
0.340 |
0.007 |
0.653
|
27 |
| SUO |
0.985
|
0.005 |
0.010 |
27 |
| RIR |
0.003 |
0.101 |
0.896
|
27 |
| LG |
0.003 |
0.995
|
0.002 |
27 |
| CN |
0.019 |
0.008 |
0.973
|
27 |
Download Excel Table
사사
본 연구는 농촌진흥청 연구사업(세부과제명: 가축유전자원 특성정보의 생산 및 관리, 세부과제번호: PJ01098402)의 지원에 의해 이루어진 것이며, 공시축의 시료 및 정보를 제공해주신 가축생명자원관리기관 담당자 여러분께 감사를 드립니다.
References
Barker JSF, Tan SG, Selvaraj, OS, Mukherjee TK 1997 Genetic variation within and relationships among populations of Asian water buffalo (Bubalus bubalis). Anim Sci 28 (1):1-13 .

Earl DA, Von-Holdt BM 2012 Structure Harvester: A website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources 4(2):359-361 .


Felsenstein J 2010 PHYLIP (Phylogeny Inference Package) Version 3.69. University of Washington, Seattle, USA .

Kong HS, Oh JD, Lee JH, Jo KJ, Sang BD, Choi CH, Kim SD, Lee SJ, Yeon SH, Jeon GJ 2006 Genetic variation and relationships of Korean native chickens and foreign breeds using 15 microsatellite markers. Asian Australas J Anim Sci 19(11):1546-1550 .


Lee HH 2010, Encyclopedia of the Chicken. Hyunchook, Seoul, Korea .

Lee PY, Yeon SH, Kim JH, Ko YG, Son JK, Lee HH, Cho CY 2011 Genetic composition of Korean native chicken populations-National scale molecular genetic evaluation based on microsatellite markers. Korean J Poult Sci 38(2): 81-87 .


Li K, Fan B, Zhao S, Peng Z, Li K, Chen Y, Moran C 2000 Analysis of diversity and genetic relationships between four Chinese indigenous pig breeds and one Australian commercial pig breed. Anim Sci 31(5):322-325 .

Martin-Burriel I, Garcia-Muro E, Zaragoza P 1999 Genetic diversity analysis of six Spanish native cattle breeds using microsatellites. Anim Sci 30(3):177-182 .

Oh JD, Kang BS, Kim HK, Park MN, Chae EJ, Seo OS, Lee HK, Jeon GJ, Kong HS 2008 Genetic relationship between populations and analysis of genetic structure in the Korean native chicken and the Endemic chicken breeds. Korean J Poult Sci 35(4):361-366 .


Park S 2000 Microsatellite Toolkit for MS Excel 97 or 2000. Personnel Communication .

Peelman LJ, Mortiaux F, Van Zeveren A, Dansercoer A, Mommens G, Coopman F, Bouquet Y, Burny A, Renaville R, Portetelle D 1998 Evaluation of the genetic variability of 23 bovine microsatellite markers in four Belgian cattle breeds. Anim Sci 29(3):161-167 .

Rosenberg NA 2004 DISTRUCT: A program for the graphical display of population structure. Mol Ecol Notes 4(1): 137-138 .


Seo JH, Oh JD, Lee JH, Seo DW, Kong HS 2015 Studies on genetic diversity and phylogenetic relationships of Korean Native Chicken using the microsatellite marker. Korean J Poult Sci 42(1):15-26 .