Article
초위성체 마커를 활용한 가축다양성정보시스템(DAD-IS) 등재 재래닭 집단의 유전적 다양성 분석
노희종1
,
김관우2
,
이진욱1
,
전다연1
,
김승창1
,
고응규3
,
문성실2
,
이현정2
,
이준헌4
,
오동엽5
,
변재현6,
조창연3,†
Genetic Diversity of Korean Native Chicken Populations in DAD-IS Database Using 25 Microsatellite Markers
Hee-Jong Roh1
,
Kwan-Woo Kim2
,
Jinwook Lee1
,
Dayeon Jeon1
,
Seung-Chang Kim1
,
Yeoung-Gyu Ko3
,
Seong-Sil Mun2
,
Hyun-Jung Lee2
,
Jun-Heon Lee4
,
Dong-Yep Oh5
,
Jae-Hyun Byeon6,
Chang-Yeon Cho3,†
Author Information & Copyright ▼
1농촌진흥청 국립축산과학원 가축유전자원센터 농업연구사
2농촌진흥청 국립축산과학원 가축유전자원센터 연구원
3농촌진흥청 국립축산과학원 가축유전자원센터 농업연구관
1Researcher, Animal Genetic Resources Center, National Institute of Animal Science, RDA, Namwon 55717, Republic of Korea
2Assistant Researcher, Animal Genetic Resources Center, National Institute of Animal Science, RDA, Namwon 55717, Republic of Korea
3Senior Researcher, Animal Genetic Resources Center, National Institute of Animal Science, RDA, Namwon 55717, Republic of Korea
4Professor, Division of Animal and Dairy Science, Chungnam National University, Daejeon 34134, Republic of Korea
5Researcher, Gyeongbuk Livestock Research Institute, Yeongju 36052, Republic of Korea
6Officer, Jeju Livestock Development Institute, Jeju 63078, Republic of Korea
© Copyright 2019, Korean Society of Poultry Science. This is an Open-Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Mar 12, 2019 ; Revised: Apr 10, 2019 ; Accepted: Apr 15, 2019
Published Online: Jun 30, 2019
ABSTRACT
A number of Korean native chicken(KNC) populations were registered in FAO (Food and Agriculture Organization) DAD-IS (Domestic Animal Diversity Information Systems, http://www.fao.org/dad-is). But there is a lack of scientific basis to prove that they are unique population of Korea. For this reason, this study was conducted to prove KNC’s uniqueness using 25 Microsatellite markers. A total of 548 chickens from 11 KNC populations (KNG, KNB, KNR, KNW, KNY, KNO, HIC, HYD, HBC, JJC, LTC) and 7 introduced populations (ARA: Araucana, RRC and RRD: Rhode Island Red C and D, LGF and LGK: White Leghorn F and K, COS and COH: Cornish brown and Cornish black) were used. Allele size per locus was decided using GeneMapper Software (v 5.0). A total of 195 alleles were observed and the range was 3 to 14 per locus. The MNA, Hexp, Hobs, PIC value within population were the highest in KNY (4.60, 0.627, 0.648, 0.563 respectively) and the lowest in HYD (1.84, 0.297, 0.286, 0.236 respectively). The results of genetic uniformity analysis suggested 15 cluster (ΔK = 66.22). Excluding JJC, the others were grouped in certain cluster with high genetic uniformity. JJC was not grouped in certain cluster but grouped in cluster 2 (44.3%), cluster 3 (17.7%) and cluster8 (19.1%). As a results of this study, we can secure a scientific basis about KNC’s uniqueness and these results can be use to basic data for the genetic evaluation and management of KNC breeds.
Keywords: Korean native chicken; genetic diversity; DAD-IS; microsatellite
서 론
우리나라에 존재하고 있던 재래닭은 일제강점기 외래품종의 도입과 한국전쟁으로 인해 그 수가 급격히 감소하였고, 1960년대 이후 근대화 과정에서 산란능력과 육질이 우수한 외래품종의 선호도가 증가함에 따라 상대적으로 경제능력이 떨어지는 재래종은 멸종위기에 처하였지만, 그 혈통을 유지해온 소수의 농가들의 노력으로 완전한 멸종은 피한 채 그 명맥을 유지할 수 있었다. 이후 유전자원이라는 개념의 보편화 이전까지 재래닭이라는 이름으로 각 농가마다 형질이 서로 다른 집단을 사육해온 결과, 다양한 집단들이 현재까지 유지될 수 있었다. 그러나 재래닭은 품종으로서 균일도가 떨어질 뿐만 아니라, 낮은 경제형질로 인해 우리나라 양계산업은 약 90% 이상 해외 여러 나라들로부터 수입해온 우수한 종자들에 의지해오고 있는 실정이다(Choi et al, 2015). 이에 따라 유전적으로 균일도가 높은 재래닭 기초집단을 조성하기 위해 국립축산과학원은 1992년부터 재래닭 수집 및 복원 사업을 진행하였고, 15년간의 노력 끝에 지난 2007년 5가지 모색(적갈색, 황갈색, 회갈색, 백색, 흑색)을 나타내는 재래닭 5개의 집단을 복원하는데 성공하였다(NIAS, 2008).
1992년 생물다양성협약(Convention on Biological Diversity; CBD) 이후 세계적으로 가축유전자원의 중요성이 점점 커짐에 따라, 분자생물학적 수준에서 각 나라의 고유한 품종임을 증명하기 위해 다양한 마커를 이용하여 해당 품종의 유전적 다양성 및 타품종과의 유연관계 등을 구명하기 위한 연구가 활발하게 진행되었고(Kaya et al., 2008; Medugorac et al., 2009; Groeneveld et al., 2010; Lenstra et al., 2012), 특히 최근 나고야 의정서 국내 발효(2017.8.)로 인해 이러한 연구를 통한 우리나라의 가축유전자원에 대해 고유종자로서의 과학적 근거를 확보함으로써 가축유전자원에 대한 주권을 확보하는데 시동이 걸리고 있다. 현재 국제식량농업기구(Food and Agriculture Organization; FAO)에서는 전 세계 가축유전자원을 체계적으로 관리하기 위해 가축다양성정보시스템(Domestic Animal Diversity Information System; DAD-IS, http://dad.fao.org)을 운영하고 있다. 이 시스템 등재는 가축유전자원의 품종을 인정하는 공식적인 국가제도가 없는 실정에서 국내 고유 가축유전자원에 대한 주권을 행사할 수 있는 유일한 기회가 되고 있다.
국립축산과학원에서는 해마다 국내 가축유전자원을 본 시스템에 등재하기 위해 지속적으로 노력을 기울이고 있으며, 복원에 성공한 5개 재래닭 집단은 물론, 수십 년 이상 보존․육성해온 자원으로 그 고유성을 인정받은 다양한 집단의 농가유래 재래닭 또한 이 시스템에 등재되어 있다. 현재 재래닭 및 외래토착종 집단을 포함하여 총 40개 집단이 이 시스템에 등재되어 있으며, 해당 집단을 보유하고 있는 농가는 관리농가로 지정이 되어 국가유전자원으로서 철저하게 관리되고 있다.
Microsatellite(MS) marker는 유전체 전체에 분포하고 있으며, 2∼6개의 단순한 염기서열이 반복되는 특징을 가지고 있고, 각 품종 및 개체간 염기서열의 반복수에서 차이가 나기 때문에 PCR 증폭 시 다양한 크기의 증폭산물이 나타난다(Cheng and Crittenden, 1994; Kaya and Yildz, 2008). 따라서 증폭산물의 크기에 따라 대립유전자가 다양하며, 공우성의 특징이 있어 이형접합체의 여부를 판단할 수 있다(Bowcock et al., 1994). 또한 세대당 돌연변이율이 10—4 이상으로 높으며(Roy et al., 1994), 개체간의 차이를 보이면서도 다음 세대로 유전되기 때문에 닭을 비롯한 다양한 동식물 품종들에 대한 연구에서 이용되고 있다(Powell et al., 1996). 2006년 이후 MS 마커를 이용한 우리나라 재래닭 집단에 대한 연구가 활발하게 진행되어 왔으며, 현재까지 국립축산과학원에서 복원한 5개 재래닭 집단, 생산성을 높이기 위해 육종해 온 우리맛닭 및 한협닭 등을 대상으로 진행한 연구들은 다수 보고되어 있지만(Oh et al., 2008; Lee et al., 2010; Lee et al., 2011; Suh et al., 2014, Seo et al., 2015), DAD-IS 등재 집단, 특히 농가에서 유래한 집단을 대상으로 진행한 연구는 부족한 실정이기에 해당 집단이 우리나라의 고유한 품종임을 증명할 수 있는 과학적인 근거가 부족하다.
따라서 본 연구에서는 DAD-IS 시스템에 등재되어 있으며, 국립축산과학원에서 복원되어 보존 및 육성 중인 재래닭 5개 집단(회갈색, 흑색, 적갈색, 백색, 황갈색)과 연산오계, 외래 도입 토착종 4품종(아라우카나, 로드아일랜드레드, 화이트레그혼, 코니시) 7개 집단 그리고 제주 재래닭 및 관리농가 유래 재래닭 4개 집단(현인, 횡성약닭, 황봉, 풍동)을 포함하여 총 18개 집단 548수를 대상으로 25개의 MS marker를 이용해 집단별 유전자형을 분석하고, 이를 토대로 유전적 다양성 및 유연관계를 구명함으로써 DAD-IS에 등재된 우리나라의 재래닭 자원에 대해 고유 종자로써의 과학적인 근거를 마련하고자 한다.
재료 및 방법
1. 공시재료 및 MS 마커
본 연구에 이용된 공시재료로는 국립축산과학원 가축유전자원센터에서 중복보존 중인 재래닭 5개 집단(회갈색, 흑색, 적갈색, 백색, 황갈색) 및 연산오계 집단과 제주 및 관리농가 유래 재래닭 4개 집단(현인, 횡성약닭, 황봉, 풍동)에 대조구로 외래 도입 토착종 4품종(아라우카나, 로드아일랜드레드, 화이트레그혼, 코니시) 7개 집단을 추가하여 총 18개 집단 548수를 이용하였으며(Table 1), 익하정맥에서 채혈한 혈액으로부터 Genomic DNA Purification Kit(Promega, Madison, Wisconsin, USA)를 이용하여 DNA를 추출하였다.
Table 1.
Description of the 18 chicken populations used in this study
| Sort |
Abbreviation |
Populations |
No. of sample |
Genetic origin |
Source |
| Imported chicken |
ARA |
Araucana |
32 |
Chile |
NIAS |
| RRC |
Rhode Island Red |
32 |
USA |
NIAS |
| RRD |
Rhode Island Red |
32 |
USA |
NIAS |
| LGF |
White Leghorn |
32 |
Italy |
NIAS |
| LGK |
White Leghorn |
32 |
Italy |
NIAS |
| COS |
Cornish Brown |
32 |
UK |
NIAS |
| COH |
Cornish Black |
32 |
UK |
NIAS |
| Korean native chicken |
KNG |
Korean Grayish-Brown |
32 |
Korea |
NIAS |
| KNB |
Korean Black |
32 |
Korea |
NIAS |
| KNR |
Korean Reddish-Brown |
32 |
Korea |
NIAS |
| KNW |
Korean White |
32 |
Korea |
NIAS |
| KNY |
Korean Yellowish-Brown |
32 |
Korea |
NIAS |
| KNO |
Korean Ogye |
32 |
Korea |
NIAS |
| HIC |
Hyun-in Black |
32 |
Korea |
Hyun-in farm |
| HYD |
Hoengseong Yakdak |
32 |
Korea |
Hoengseong farm |
| HBC |
Hwangbong |
30 |
Korea |
Hwangbong farm |
| JJC |
Jeju Native Chicken |
23 |
Korea |
JLDI |
| LTC |
Long Tail Chicken |
15 |
Korea |
Poongdong farm |
Download Excel Table
집단별 다양성 분석을 위해 사용한 MS marker는 ISAG/ FAO(FAO, 2004)에서 권고하고 있는 MS marker 10종(ADL0278, LEI0094, LEI0166, MCW0016, MCW0037, MCW0078. MCW0111, MCW0206, MCW0295, MCW0330)과 Roslin Bioinfomatics Group에서 제공하는 ArkDB database website를 참고하여 다양성이 높게 보고된 15종(ADL0176, ADL0267, LEI0092, LEI0096, LEI0099, LEI0135, LEI0209, MCW0145, MCW0193, MCW0214, MCW0233, MCW0240, MCW0252, MCW0301, MCW0322)의 마커를 선별하여 총 25종을 이용하였다.
2. PCR 및 유전자형 분석
추출된 DNA는 UV Spectrophotometer (NanoDrop NA-1000; Thermo Scientific, Waltham, Massachusetts, USA)을 이용해 농도를 측정한 후, 최종 10 ng/μL가 되도록 증류수에 희석하여 PCR 증폭을 위한 주형으로 사용하였다. 각 마커별 증폭산물의 크기와 형광표지 등을 고려하여 2∼6개씩 Multiplex 조합을 만든 후, DNA 중합효소, dNTP, KCl, MgCl2 등이 포함되어 있는 AccuPowerTM Negative dye PCR PreMix (Bioneer, Daejeon, Korea)를 이용하여 주형 DNA 2 μL, primer는 각각의 multiplex 조합별로 조건에 맞게 0.4 μL∼2.6 μL(2 pmole/μL)을 첨가하고, 최종부피가 20 μL가 되도록 증류수를 첨가한 후, multiplex PCR을 수행하였다. PCR 반응은 GeneAmp PCR system 9700(Applied Biosystems, Foster, California, USA)을 이용하여 수행하였으며, 95℃에서 5분간 pre-denaturation, 95℃에서 60초간 denaturation, 각 조합에 따라 55∼62℃에서 45초간 annealing, 72℃에서 60초간 extension을 35회 반복한 후 72℃에서 30분 동안 final extension을 실시하였다.
증폭된 PCR 산물은 Hi-Di Foramide(Applied Biosystems, USA)와 GeneScan 500Liz Size Standard(Applied Biosystems, USA)를 혼합하여 10분 동안 95℃에서 denaturation 시킨 후 즉시 얼음에 넣어 상태를 유지하게 한 다음 자동 염기 서열 분석 장치인 ABI PRISM 3130XL Genetic Analyzer(Applied Biosystems, USA)에 장착하여 POP 7 polymer(Applied Biosystems, USA)상에서 모세관 전기영동을 통해 분석하였다. 각 마커별 대립유전자들의 크기는 GeneMapper version 5.0 software(Applied Biosystems, USA)를 이용하여 결정하였고, 크기가 결정된 좌위들은 공시된 개체별로 Microsoft Excel 2016(Microsoft, Redmond, Washington, USA)을 이용하여 자료를 취합한 후 각 통계분석 방법에 적용하였다.
3. 통계분석
크기가 결정된 마커별 대립유전자들은 MS tool Kit(Park, 2000)을 이용하여 마커 및 집단별 관측이형접합도(Hobs), 기대이형접합도(Hexp), 다형정보지수(PIC) 등을 계산하였다. 집단간 유전 거리 추정을 위해서는 DISPAN(Ota, 1993)을 이용하여 Nei 등(1983)의 방법에 따라 DA 유전거리를 산출하였으며, 이를 토대로 PHYLIP program pakage(http://evolution.genetics.washington.edu/phylip.html, Felesenstein, 2010)을 이용하여 Neighbor-Joining phylogenetic tree를 작성하였고, TreeView 1.6(ftp://eeb.lu.lv/pub/GIS/datu_analiize/TreeView/treeview_manual.html, Roderic, 1996)를 이용하여 도식화였다.
집단의 유전적 균일도 추정은 STRUCTURE software (Pritchard et al., 2000)을 이용하였으며, 구분 가능한 집단 수(K값)을 추정하기 위해서 K를 2부터 18까지 설정하였고, 각 값에 대한 평균 추정치 Pr(X│K)와 표준편차를 계산하고, burn-in 20,000회, MCMC(markov chain Monte Carlo)반복수 100,000회, 이를 10회 반복하여 계산된 결과를 Evanno 등(2005)의 방법이 적용된 STRUCTURE HARVESTER (Earl and von-Holdt, 2012)를 사용하여 ΔK 값을 계산하여 최적의 K값을 결정하였으며, CLUMMP(Jakobsson and Rosengerg, 2007)를 이용해 10회 반복하여 나온 최적의 K값에 대한 cluster별 유전적 균일도를 계산하였고, 이후 각 cluster에 대한 집단의 실제 분포를 DISTRUCT(Rosenberg, 2004)를 이용하여 도식화 하였다.
결과 및 고찰
1. 마커 및 집단별 유전적 다양성
25개 MS마커를 이용하여 11개 재래닭 집단, 7개 외래 도입 토착종 집단을 포함하여 총 18개 집단 548수에 대한 마커별 대립유전자 수(NA), 기대이형접합도(Hexp), 관측이형접합도(Hobs), 다형정보지수(PIC)를 Table 2에 제시하였다. 마커별 대립유전자 수의 경우 3∼14개의 범위로 총 195개의 대립유전자가 나타났으며, 마커별 평균 7.80개의 대립유전자를 보유하고 있는 것으로 확인되었다. Hexp와 PIC의 경우 MCW0145에서 각각 0.646, 0.569, Hobs의 경우 ADL0278에서 0.773으로 가장 높게 나타났으며, 반대로 MCW0078에서 는 Hexp, Hobs, PIC 각각 0.263, 0.291, 0.217로 가장 낮은 수치를 보이고 있었다. 한편, 다형성이 높게 나타난 MCW0145의 경우 우리나라 재래닭 및 외래 도입 토착종 집단을 대상으로 진행한 여러 논문(Kong et al., 2006; Seo et al., 2013; Suh et al., 2014)에서도 높은 다형성을 나타내고 있는 것으로 보고되었으며, 다형성이 낮게 나타난 MCW0078의 경우에도 우리나라 재래닭 집단에서뿐만 아니라, 다른 여러 국가의 닭 집단에서도 낮은 다형성을 나타내고 있는 것으로 보고되었다(Tadano et al., 2007; Ding et al., 2010; Abebe et al., 2015; Long et al., 2017).
Table 2.
The statistical analysis of heterozygosity, polymorphism information content using 25 MS markers
| Locus |
NA |
Hexp |
Hobs |
PIC |
| ADL0176 |
10 |
0.559±0.042 |
0.538±0.058 |
0.486±0.039 |
| ADL0267 |
10 |
0.440±0.056 |
0.368±0.058 |
0.379±0.050 |
| ADL0278 |
8 |
0.629±0.019 |
0.773±0.052 |
0.553±0.022 |
| LEI0092 |
8 |
0.469±0.055 |
0.424±0.058 |
0.408±0.049 |
| LEI0094 |
13 |
0.533±0.043 |
0.546±0.045 |
0.470±0.041 |
| LEI0096 |
8 |
0.541±0.042 |
0.555±0.050 |
0.477±0.039 |
| LEI0099 |
9 |
0.414±0.045 |
0.396±0.050 |
0.354±0.039 |
| LEI0135 |
7 |
0.500±0.044 |
0.485±0.046 |
0.424±0.041 |
| LEI0166 |
4 |
0.370±0.050 |
0.359±0.052 |
0.316±0.044 |
| LEI0209 |
11 |
0.602±0.043 |
0.605±0.046 |
0.544±0.042 |
| MCW0016 |
5 |
0.438±0.060 |
0.423±0.062 |
0.385±0.054 |
| MCW0037 |
4 |
0.486±0.040 |
0.499±0.044 |
0.408±0.035 |
| MCW0078 |
4 |
0.263±0.045 |
0.291±0.053 |
0.217±0.037 |
| MCW0111 |
7 |
0.518±0.029 |
0.510±0.037 |
0.437±0.027 |
| MCW0145 |
8 |
0.646±0.021 |
0.644±0.020 |
0.569±0.026 |
| MCW0193 |
10 |
0.549±0.046 |
0.569±0.045 |
0.491±0.043 |
| MCW0206 |
7 |
0.499±0.033 |
0.504±0.044 |
0.428±0.029 |
| MCW0214 |
10 |
0.556±0.042 |
0.556±0.043 |
0.494±0.041 |
| MCW0233 |
4 |
0.437±0.054 |
0.418±0.053 |
0.369±0.047 |
| MCW0240 |
14 |
0.554±0.052 |
0.539±0.050 |
0.494±0.049 |
| MCW0252 |
8 |
0.561±0.028 |
0.543±0.045 |
0.488±0.030 |
| MCW0295 |
8 |
0.513±0.030 |
0.494±0.044 |
0.436±0.029 |
| MCW0301 |
9 |
0.533±0.055 |
0.554±0.060 |
0.473±0.050 |
| MCW0322 |
3 |
0.545±0.032 |
0.385±0.049 |
0.335±0.040 |
| MCW0330 |
6 |
0.405±0.048 |
0.583±0.040 |
0.460±0.032 |
Download Excel Table
Table 3에서는 공시된 18개의 집단별 평균대립유전자수(MNA), 기대이형접합도(Hexp), 관측이형접합도(Hobs), 다형정보지수(PIC)를 제시하였다. 분석 결과, 모든 수치가 황갈색재래종(KNY) 집단(MNA: 4.60, Hexp: 0.627, Hobs: 0.643, PIC: 0.563)에서 가장 높게, 횡성약닭(HYD) 집단(MNA: 1.84, Hexp: 0.297, Hobs: 0.286, PIC: 0.236)에서 가장 낮은 것으로 확인되었다. 특히 횡성약닭(HYD) 집단의 경우, 모든 지표에서 현저하게 낮은 다양성 수치를 나타내었는데, 이는 다른 집단과는 달리 매우 소수의 개체수를 가지고 그 혈통을 유지하고 있기 때문인 것으로 사료된다. 또한 산란계에 속하는 화이트레그혼 두 집단(LGF, LGK)과 아라우카나(ARA) 집단의 경우 다른 집단에 비해 상대적으로 낮은 다양성을 보이고 있는데, 이는 타집단에 비해 산란능력 발현을 위해서 유전적 고정화 작업이 강력하게 이루어졌기 때문인 것으로 사료된다.
Table 3.
Genetic diversity parameters in 9 chicken populations
| Population |
Sample size |
Marker |
MNA |
Hexp |
Hobs |
PIC |
| ARA |
32 |
25 |
2.92±1.35 |
0.424±0.050 |
0.433±0.018 |
0.372±0.045 |
| RRC |
32 |
25 |
2.76±0.88 |
0.487±0.037 |
0.489±0.018 |
0.411±0.033 |
| RRD |
32 |
25 |
2.80±0.82 |
0.501±0.034 |
0.523±0.018 |
0.426±0.031 |
| LGF |
32 |
25 |
2.96±0.93 |
0.419±0.044 |
0.432±0.018 |
0.353±0.037 |
| LGK |
32 |
25 |
3.00±1.12 |
0.431±0.042 |
0.440±0.018 |
0.375±0.038 |
| COS |
32 |
25 |
3.24±0.93 |
0.528±0.028 |
0.525±0.018 |
0.452±0.026 |
| COH |
32 |
25 |
3.36±1.04 |
0.503±0.037 |
0.493±0.018 |
0.439±0.033 |
| KNG |
32 |
25 |
3.08±1.00 |
0.478±0.031 |
0.509±0.018 |
0.410±0.028 |
| KNB |
32 |
25 |
4.08±1.26 |
0.585±0.028 |
0.595±0.017 |
0.520±0.028 |
| KNR |
32 |
25 |
4.36±1.41 |
0.608±0.030 |
0.625±0.017 |
0.548±0.028 |
| KNW |
32 |
25 |
3.76±1.36 |
0.570±0.030 |
0.566±0.018 |
0.497±0.031 |
| KNY |
32 |
25 |
4.60±1.58 |
0.627±0.025 |
0.643±0.017 |
0.563±0.027 |
| KNO |
32 |
25 |
3.40±1.26 |
0.507±0.040 |
0.499±0.018 |
0.445±0.036 |
| HIC |
32 |
25 |
4.16±1.43 |
0.581±0.032 |
0.555±0.018 |
0.515±0.031 |
| HYD |
32 |
25 |
1.84±0.69 |
0.297±0.042 |
0.286±0.016 |
0.236±0.033 |
| HBC |
30 |
25 |
2.40±0.82 |
0.423±0.049 |
0.415±0.018 |
0.351±0.042 |
| JJC |
23 |
25 |
3.96±1.06 |
0.581±0.024 |
0.573±0.021 |
0.513±0.024 |
| LTC |
15 |
25 |
2.96±1.31 |
0.496±0.044 |
0.444±0.026 |
0.420±0.041 |
Download Excel Table
2. 집단 간 유전거리 및 유연관계
공시된 18개 집단 간 유전거리 및 유연관계를 추정하기 위해 마커별 대립유전자의 빈도를 바탕으로 DISPAN(Ota, 1993)을 통해 계산된 집단간 DA 유전거리(Nei et al., 1983)를 Table 4에 제시하였다. 분석 결과, 횡성약닭(HYD)과 화이트레그혼F(LGF) 집단 사이에서 0.675의 가장 먼 유전거리를 나타냈으며, 같은 품종인 로드아일랜드레드 두 집단(RRC, RRD) 사이에서 0.027의 가장 가까운 유전거리를 보였고, 역시 같은 품종인 화이트레그혼 두 집단(LGF, LGK) 사이에서도 0.092의 매우 가까운 유전거리를 보였다. 한편, 코니시 두 집단(COS, COH) 사이에서는 같은 품종임에도 불구하고, 0.313의 비교적 먼 유전거리를 나타내고 있는데, 이는 두 집단이 유전적 차이로 인해 갈색(COS)과 흑색(COH)으로 구분되는 집단이기 때문인 것으로 사료된다. 재래닭 집단간의 유전거리를 살펴보면 횡성약닭(HYD)과 긴꼬리닭(LTC) 집단 사이에서 0.611로 가장 먼, 적갈색재래종(KNR)과 황갈색재래종(KNY) 집단에서 0.187로 가장 가까운 유전거리를 나타내고 있었으며, 동일한 집단을 공시재료로 이용한 Lee et al.(2011), Suh et al.(2014)의 연구 결과에서도 적갈색재래종(KNR)과 황갈색재래종(KNY) 집단 사이에서 굉장히 가까운 유전거리를 형성하고 있는 것으로 보고되었다.
Table 4.
Matrix of DA genetic distances observed among 18 chicken populations
|
|
ARA |
RRC |
RRD |
LGF |
LGK |
COS |
COH |
KNG |
KNB |
KNR |
KNW |
KNY |
KNO |
HIC |
HYD |
HBC |
JJC |
| RRC |
0.373 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| RRD |
0.350 |
0.027 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| LGF |
0.434 |
0.514 |
0.515 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
| LGK |
0.400 |
0.486 |
0.493 |
0.092 |
|
|
|
|
|
|
|
|
|
|
|
|
|
| COS |
0.446 |
0.336 |
0.318 |
0.457 |
0.437 |
|
|
|
|
|
|
|
|
|
|
|
|
| COH |
0.372 |
0.325 |
0.323 |
0.470 |
0.444 |
0.313 |
|
|
|
|
|
|
|
|
|
|
|
| KNG |
0.414 |
0.280 |
0.296 |
0.518 |
0.511 |
0.426 |
0.269 |
|
|
|
|
|
|
|
|
|
|
| KNB |
0.376 |
0.296 |
0.281 |
0.450 |
0.417 |
0.341 |
0.255 |
0.304 |
|
|
|
|
|
|
|
|
|
| KNR |
0.293 |
0.292 |
0.281 |
0.454 |
0.431 |
0.333 |
0.244 |
0.276 |
0.206 |
|
|
|
|
|
|
|
|
| KNW |
0.369 |
0.323 |
0.326 |
0.425 |
0.381 |
0.386 |
0.307 |
0.296 |
0.250 |
0.247 |
|
|
|
|
|
|
|
| KNY |
0.359 |
0.352 |
0.331 |
0.481 |
0.466 |
0.362 |
0.291 |
0.252 |
0.248 |
0.187 |
0.230 |
|
|
|
|
|
|
| KNO |
0.437 |
0.360 |
0.373 |
0.438 |
0.430 |
0.347 |
0.263 |
0.386 |
0.267 |
0.253 |
0.374 |
0.315 |
|
|
|
|
|
| HIC |
0.371 |
0.326 |
0.328 |
0.472 |
0.463 |
0.351 |
0.214 |
0.275 |
0.218 |
0.210 |
0.271 |
0.237 |
0.251 |
|
|
|
|
| HYD |
0.498 |
0.465 |
0.475 |
0.675 |
0.637 |
0.523 |
0.465 |
0.506 |
0.411 |
0.386 |
0.433 |
0.433 |
0.411 |
0.398 |
|
|
|
| HBC |
0.529 |
0.482 |
0.474 |
0.549 |
0.516 |
0.435 |
0.453 |
0.448 |
0.351 |
0.319 |
0.408 |
0.336 |
0.424 |
0.379 |
0.521 |
|
|
| JJC |
0.375 |
0.305 |
0.302 |
0.425 |
0.418 |
0.349 |
0.342 |
0.324 |
0.259 |
0.280 |
0.287 |
0.221 |
0.309 |
0.273 |
0.535 |
0.441 |
|
| LTC |
0.489 |
0.410 |
0.404 |
0.537 |
0.491 |
0.491 |
0.420 |
0.381 |
0.411 |
0.334 |
0.411 |
0.378 |
0.507 |
0.443 |
0.611 |
0.465 |
0.390 |
Download Excel Table
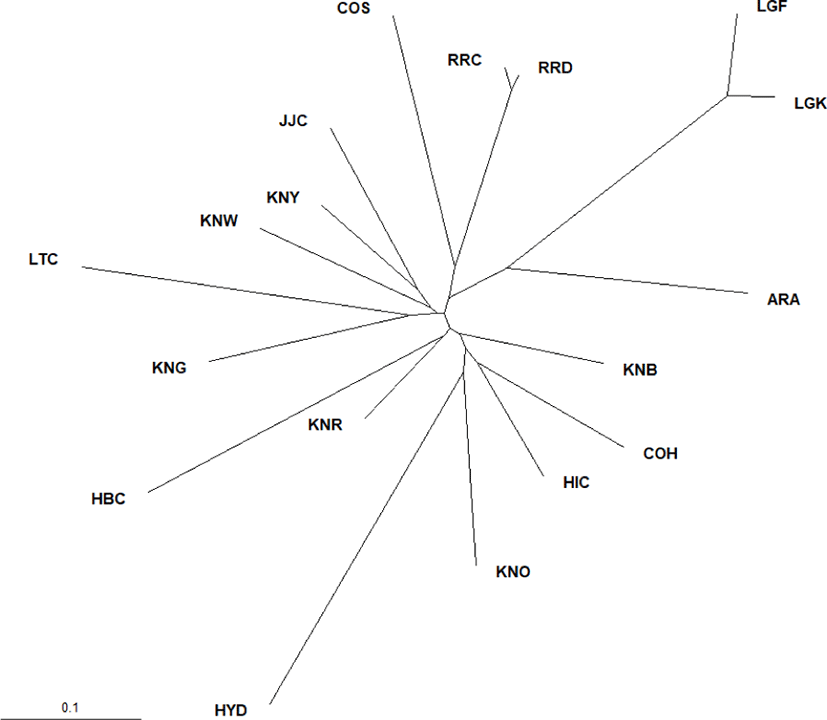
각 집단간 추정된 유전거리를 바탕으로 PHYLIP program package을 이용하여 Neighbor-joining(Saitou and Nei, 1987)의 방법으로 작성한 집단간 Phylogenetic tree를 Fig. 1에 제 시하였다. 전반적으로 화이트레그혼 두 집단(LGF, LGK)이 별도의 가지를 형성하는 것으로 나타났으며, 청란을 낳는다고 보고되어 있는(Wang et al., 2013) 아라우카나 집단(ARA)이 산란계인 화이트레그혼 두 집단과 같은 군집을 형성하고 있는 것을 확인할 수 있었다. 또한, 코니시 두 집단(COS, COH)는 같은 품종임에도 불구하고, 서로 다른 가지를 형성하고 있는 것을 다시 한 번 확인할 수 있었다. 한편, 흑색재래종(KNB), 현인흑계(HIC), 연산오계(KNO) 집단의 경우 흑색코니시(COH) 집단과 같은 군집을 형성하고 있는 것으로 나타났는데, 이는 4개의 집단 모두 흑색의 모색을 가지고 있기 때문인 것으로 사료되며, 전반적으로 각 집단이 비교적 확연하게 구분되는 것을 확인할 수 있었다.
Fig. 1.
Neighbor-Joining Phylogenetic tree showing genetic relationships among 18 chicken populations based on DA distances.
Download Original Figure
3. 집단의 균일도
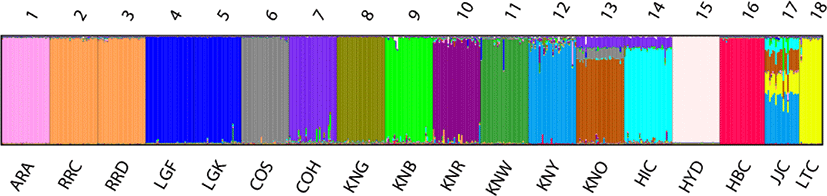
공시된 18개 집단이 유전적으로 몇 개의 군락으로 구분하는 것이 적절한지를 추론하기 위해 Bayesian clustering법이 적용된 STRUCTURE software를 이용하여 K값을 2부터 18까지 설정한 후, 10회 반복하여 분석한 결과를 바탕으로 STRUCTURE HARVESTER를 이용해 Evanno et al.(2005)의 방법으로 최적의 K값(ΔK)을 계산한 결과 K=15에서 가장 높은 ΔK(66.22)이 나타나는 것을 확인할 수 있었다(Table 5). 최적의 K값으로 나온 15는 본 연구에서 공시된 18개의 집단이 25개의 MS마커에서 나온 대립유전자의 빈도를 바탕으로 구분하였을 때 15개의 집단으로 구분하는 것이 유전적으로 가장 적합하다는 것을 의미한다. Table 6에서는 15개의 군락으로 구분했을 때의 집단별 유전적 균일도를 나타내었으며, Fig. 2에서는 집단별 군락 형태를 DISTRUCT Software(Rosenberg, 2004)를 이용하여 보기 쉽게 도식화한 결과를 제시하였다. 18개의 집단 중 14개의 집단에서 90%이상의 높은 유전적 균일도를 나타내었으며, 황갈색재래종(KNY)과 현인흑계(HIC) 집단에서도 각각 88.9%, 83.9%의 비교적 높은 균일도를 형성하며 독립적인 군락을 형성하고 있었다. 앞서 제시했던 유전거리 분석결과와 마찬가지로 같은 품종인 화이트레그혼 두 집단(LGK, LGF)과 로드아일랜드레드 두 집단(RRC, RRD)는 각각 12번 군락과 4번 군락에서 매우 높은 균일도를 형성하고 있는 것으로 나타났지만, 코니시 두 집단(COS, COH)의 경우 각각 10번 군락과 14번 군락에서 독립적인 집단으로 구분되는 것을 확인할 수 있었다. 한편, 연산오계(KNO) 집단의 경우 8번 군락에서 다소 낮은 76.3%의 균일도를 보이고 있었으나, 본 군락에서 가장 높은 균일도를 형성하고 있는 것으로 보아 독립적인 집단으로 구분하는 것이 타당하다는 결론을 내릴 수 있었으며, 이는 Suh 등(2014)의 연구 결과와 비슷한 것으로 나타났다. 반면, 제주재래닭(JJC) 집단의 경우 독립적인 군락을 형성하지 못하고, 황갈색재래종(KNY) 집단이 속해 있는 2번 군락에서 가장 높은 44.3%의 균일도를 보이고 있었으며, 3번 군락(17.7%)과 8번 군락(19.1%)에도 일부 포함되어 있는 것으로 나타났다. 이를 통해 제주 재래닭 집단은 낮은 균일도로 인해 독립적인 집단으로 구분하는 것이 어려울 것으로 판단되며, 이는 사육 과정 시 일부 개체에서 타 집단과의 교잡이 일어났기 때문인 것으로 보인다. 따라서 추후 본 집단을 독립적인 집단으로 구분하기 위해 철저한 개체 식별 및 계획 교배를 통하여 지속적인 계통 조성 과정이 필요할 것으로 사료된다.
Table 5.
The mean likelihoods of models and standard deviation, and Delta K value using Evanno methods (2005)
| K |
Repeats |
Mean LnP(K) |
Stdev LnP(K) |
Ln'(K) |
|Ln''(K)| |
Delta K |
| 2 |
10 |
−38,032.86 |
136.59 |
NA |
NA |
NA |
| 3 |
10 |
−36,069.40 |
86.64 |
1,963.46 |
356.02 |
4.11 |
| 4 |
10 |
−34,461.96 |
148.82 |
1,607.44 |
603.31 |
4.05 |
| 5 |
10 |
−33,457.83 |
1,141.03 |
1,004.13 |
314.80 |
0.28 |
| 6 |
10 |
−32,138.90 |
172.48 |
1,318.93 |
148.16 |
0.86 |
| 7 |
10 |
−30,968.13 |
98.31 |
1,170.77 |
203.85 |
2.07 |
| 8 |
10 |
−30,001.21 |
197.36 |
966.92 |
291.76 |
1.48 |
| 9 |
10 |
−29,326.05 |
284.63 |
675.16 |
143.19 |
0.50 |
| 10 |
10 |
−28,507.70 |
130.32 |
818.35 |
356.90 |
2.74 |
| 11 |
10 |
−28,046.25 |
217.24 |
461.45 |
296.10 |
1.36 |
| 12 |
10 |
−27,288.70 |
136.85 |
757.55 |
275.49 |
2.01 |
| 13 |
10 |
−26,806.64 |
122.59 |
482.06 |
198.64 |
1.62 |
| 14 |
10 |
−26,523.22 |
259.54 |
283.42 |
532.71 |
2.05 |
| 15 |
10 |
−25,707.09 |
122.09 |
816.13 |
8,084.73 |
66.22
|
| 16 |
10 |
−32,975.69 |
2,4291.27 |
−7,268.60 |
13,542.77 |
0.56 |
| 17 |
10 |
−26,701.52 |
4,429.49 |
6,274.17 |
4871.03 |
1.10 |
| 18 |
10 |
−25,298.38 |
110.35 |
1,403.14 |
NA |
NA |
Download Excel Table
Table 6.
Population of membership of each 18 chicken populations genotypes with 25 microsatellite marker in the 15 inferred clusters using STRUCTURE analysis
| POP |
Inferred cluster |
N |
| 1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
11 |
12 |
13 |
14 |
15 |
| ARA |
0.001 |
0.001 |
0.001 |
0.002 |
0.001 |
0.002 |
0.979
|
0.002 |
0.001 |
0.002 |
0.001 |
0.002 |
0.002 |
0.002 |
0.001 |
32 |
| RRC |
0.002 |
0.002 |
0.002 |
0.972
|
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.001 |
0.003 |
0.003 |
0.002 |
32 |
| RRD |
0.002 |
0.002 |
0.002 |
0.969
|
0.002 |
0.002 |
0.003 |
0.002 |
0.002 |
0.003 |
0.003 |
0.002 |
0.002 |
0.003 |
0.002 |
32 |
| LGF |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.002 |
0.984
|
0.001 |
0.001 |
0.001 |
32 |
| LGK |
0.002 |
0.002 |
0.002 |
0.001 |
0.006 |
0.002 |
0.002 |
0.002 |
0.002 |
0.001 |
0.002 |
0.971
|
0.002 |
0.002 |
0.001 |
32 |
| COS |
0.002 |
0.002 |
0.001 |
0.005 |
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.966
|
0.002 |
0.002 |
0.002 |
0.005 |
0.002 |
32 |
| COH |
0.002 |
0.003 |
0.002 |
0.002 |
0.003 |
0.005 |
0.002 |
0.004 |
0.004 |
0.005 |
0.020 |
0.002 |
0.007 |
0.936
|
0.002 |
32 |
| KNG |
0.002 |
0.003 |
0.002 |
0.004 |
0.003 |
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.002 |
0.001 |
0.969
|
0.003 |
0.001 |
32 |
| KNB |
0.004 |
0.004 |
0.002 |
0.005 |
0.004 |
0.007 |
0.003 |
0.005 |
0.008 |
0.003 |
0.935
|
0.003 |
0.004 |
0.006 |
0.009 |
32 |
| KNR |
0.004 |
0.014 |
0.006 |
0.015 |
0.006 |
0.909
|
0.005 |
0.006 |
0.005 |
0.004 |
0.006 |
0.005 |
0.005 |
0.005 |
0.005 |
32 |
| KNW |
0.003 |
0.004 |
0.002 |
0.004 |
0.940
|
0.004 |
0.004 |
0.003 |
0.003 |
0.002 |
0.005 |
0.017 |
0.004 |
0.003 |
0.003 |
32 |
| KNY |
0.010 |
0.889
|
0.004 |
0.005 |
0.016 |
0.015 |
0.010 |
0.006 |
0.006 |
0.006 |
0.007 |
0.003 |
0.010 |
0.009 |
0.006 |
32 |
| KNO |
0.003 |
0.005 |
0.002 |
0.003 |
0.002 |
0.006 |
0.003 |
0.763
|
0.006 |
0.090 |
0.006 |
0.006 |
0.003 |
0.098 |
0.005 |
32 |
| HIC |
0.003 |
0.007 |
0.003 |
0.003 |
0.006 |
0.007 |
0.003 |
0.006 |
0.839
|
0.003 |
0.007 |
0.002 |
0.008 |
0.097 |
0.006 |
32 |
| HYD |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.986
|
32 |
| HBC |
0.982
|
0.002 |
0.001 |
0.001 |
0.001 |
0.002 |
0.001 |
0.002 |
0.002 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
0.001 |
30 |
| JJC |
0.003 |
0.443
|
0.177 |
0.010 |
0.005 |
0.005 |
0.008 |
0.191 |
0.105 |
0.005 |
0.032 |
0.006 |
0.005 |
0.004 |
0.003 |
23 |
| LTC |
0.003 |
0.003 |
0.955
|
0.004 |
0.002 |
0.005 |
0.002 |
0.001 |
0.006 |
0.002 |
0.002 |
0.002 |
0.004 |
0.008 |
0.002 |
15 |
Download Excel Table
Fig. 2.
Clustering assignment of the 18 chicken populations obtained by STRUCTURE analysis (K=15).
Download Original Figure
적 요
본 연구는 세계식량농업기구(FAO) 가축다양성정보시스템(DAD-IS)에 등재되어 있는 우리나라 재래닭 집단의 유전적 다양성 및 외래품종과의 차별성을 분석하기 위해 25개의 초위성체(MS) 마커를 이용하여 총 18개 집단 548수의 유전자형을 분석하였고, 이를 토대로 기대(Hexp) 및 관측이형접합도(Hobs), 다형정보지수(PIC), 유전거리, 유전적 균일도 등을 계산하였다. 마커별 다형성 분석 결과, 총 195개의 대립유전자가 나타났으며, Hexp와 PIC의 경우 MCW0145에서 각각 0.646, 0.569로 가장 높았으며, Hobs의 경우 ADL0278에서 0.773으로 가장 높은 수치를 보이고 있었던 반면, MCW0078에서는 Hexp, Hobs, PIC가 각각 0.263, 0.291, 0.217로 가장 낮은 것을 확인할 수 있었다. 집단간 다양성 분석 결과로는 MNA, Hexp, Hobs, PIC 모두 황갈색재래종(KNY) 집단(각각 4.60, 0.627, 0.643, 0.563)에서 가장 높게, 횡성약닭(HYD) 집단(각각 1.84, 0.297, 0.286, 0.236)에서 가장 낮게 나타났다.
대립유전자형의 빈도를 바탕으로 계산된 18개 품종간의 DA 유전거리 분석 결과, 횡성약닭(HYD)와 화이트레그혼F(LGF) 집단 사이에서 0.675로 가장 먼 유전거리를 형성하고 있었으며, 같은 품종인 로드아일랜드레드 두 집단(RRC, RRD) 사이에서 0.027로 가장 가까운 유전거리를 보였다. 한편, 같은 품종임에도 불구하고, 코니시 두 집단(COS, COH) 사이에서는 0.313의 비교적 먼 유전거리를 나타내고 있었다. 집단의 실제 구조를 확인하기 위한 집단별 균일도 분석 결과, K=15에서 최적의 K값(ΔK:66.22)을 얻을 수 있었으며, 18개의 집단 중 14개의 집단에서 90% 이상의 높은 유전적 균일도를 나타내며 독립적인 군락을 형성하고 있었다. 또한, 황갈색재래종(KNY), 현인흑계(HIC), 연산오계(KNO) 집단에서도 각각 88.9%, 83.9%, 76.3%로 독립적인 군락을 형성하고 있는 것을 확인할 수 있었다. 반면, 제주재래닭(JJC)의 경우 독립적인 군락을 형성하지 못하고, 황갈색재래종(KNY) 집단이 속해 있는 2번 군락에서 가장 높은 44.3%의 균일도를 보이고 있었으며, 3번 군락(17.7%)과 8번 군락(19.1%)에도 일부 포함되어 있는 것으로 보아 집단 조성 과정에 있어 타집단과의 교잡이 일어났을 것으로 추정되며, 독립적인 집단으로 구분하는 것이 어렵기 때문에 추후 개체식별을 통한 지속적인 계획교배를 실시하여 유전적 고정화 작업이 이루어질 필요성이 있을 것으로 판단된다.
이상의 결과로 DAD-IS에 등재되어 있는 우리나라 재래닭 집단이 외래 토착종 집단과 확연하게 구분이 되며, 각 재래닭 집단간에도 비교적 뚜렷하게 구분되는 것을 확인함으로써, 고유 종자로서의 과학적인 근거를 확보할 수 있었으며, 추후 재래닭 유전자원에 대한 국가 수준의 관리 및 평가를 통해 다양한 육종 소재로 이용할 수 있는 기초자료로써 활용될 수 있을 것으로 보인다.
사 사
본 연구는 농촌진흥청 연구사업(세부과제명: 가축유전자원 특성정보의 생산 및 관리, 세부과제번호: PJ01098402)의 지원에 의해 이루어진 것이며, 공시축의 시료 및 정보를 제공해주신 가축생명자원관리기관 담당자와 재래닭 현지보존 관리농가에게 깊은 감사를 드립니다.
REFERENCES
Abebe AS, Mikko S, Johansson AM 2015 Genetic diversity of five local Swedish chicken breeds detected by microsatellite markers. PLoS One 10(4):e0120580.

Bowcock AM, Ruiz-Linares A, Tomfohrde J, Minch E, Kidd JR, Cavalli-Sforza LL 1994 High resolution of human evolutionary trees with polymorphic microsatellites. Nature 368(6470):455-457.

Cheng HH, Crittenden LB 1994 Microsatellite markers for genetic mapping in the chicken. Poult Sci 73(4): 539-546.

Ding FX, Zhang GX, Wang JY, Li Y, Zhang LJ, Wei Y, Wang HH, Zhang L, Hou QR 2009 Genetic diversity of a Chinese native chicken breed, Bian chicken, based on twenty-nine microsatellite markers. Asian-Australasian J Anim Sci 23(2):154-161.

Earl DA, Von-Holdt BM 2012 STRUCTURE HARVESTER: A website and program for visualizing STRUCTURE output and implementing the Evanno method. Conserv Genet Resour 4(2):359-361.


FAO 2004 Secondary Guidelines. Measurement of Domestic Animal Diversity (MoDAD): Recommended Microsatellite Markers. Available at:
http://fao.org/dad-is.

Felsenstein J 2010 PHYLIP (Phylogeny Inference Package) Version 3.69. University of Washington, Seattle, USA.

Kong HS, Oh JD, Lee JH, Jo KJ, Sang BD, Choi CH, Kim SD, Lee SJ, Yeon SH, Jeon GJ 2006 Genetic variation and relationships of Korean native chickens and foreign breeds using 15 microsatellite markers. Asian-Australasian J Anim Sci 19(11):1546-1550.


Lee KW, Oh JD, Lee JA, Cho KH, Nam IS, Lee JH, Seo OK, Jeon GJ, Lee HK, Kong HS 2010 Estimation of genetic characteristic and cumulative power of discrimination using the microsatellite markers in Korean native chicken. Korean J Poult Sci 37(1):81-87.

Lee PY, Yeon SH, Kim JH, Ko YG, Son JK, Lee HH, Cho CH 2011 Genetic composition of Korean native chicken populations-National scale molecular genetic evaluation based on microsatellite markers. Korean J Poult Sci 38(2): 81-87.

Lenstra JA, Groeneveld LF, Eding H, Kantanen J, Williams JL, Taberlet P, Nicolazzi EL, Solkner J, Simianer H, Ciani E 2012 Molecular tools and analytical approaches for the characterization of farm animal genetic diversity. Anim Genet 43(5):483-502.

Long A, Qing Y, Gu T, Zhu Q, Liu Y, Wang Y, Yin H, Shu G, Zhang Y, Lai S 2017 Genetic variation of nine chicken breeds collected from different altitudes revealed by microsatellites. J Poult Sci 54(1):18-25.

Medugorac I, Medugorac A, Russ I, Veit-Kensch C, Taberlet P, Luntz B, Mix HM, Foerster M 2009 Genetic diversity of European cattle breeds highlights the conservation value of traditional unselected breeds with high effective population size. Mol Ecol 18(16):3394-3410.

Nei M, Tajima F, Tateno Y 1983 Accuracy of estimated phylogenetic trees from molecular data. J Mol Evol 19(2): 153-170.

NIAS 2008 Korean Native Chicken Certification Standard Institution Research. Suwon, Korea

Oh JD, Kang BS, Kim HK, Park MN, Chae EJ, Seo OS, Lee HK, Jeon GJ, Kong HS 2009 Genetic relationship between populations and analysis of genetic structure in the Korean native chicken and the endemic chicken breeds. Korean J Poult Sci 35(4):361-366.

Ota T 1993 DISPAN: Genetic Distance and Phylogeny Analysis. Pennsylvania State University, PA, USA.

Page, Roderic DM 1996 TreeView: An application to display phylogenetic trees on personal computers. Bioinform 12(4): 357-358.


Park S 2000 Microsatellite Toolkit for MS Excel 97 or 2000. Personnel Communication.

Powell W, Machray GC, Provan J 1996 Polymorphism revealed by simple sequence repeats. Trends Plant Sci 1(7):215-222.


Pritchard JK, Stephens M, Donnelly P 2000 Inference of population structure using multilocus genotype data. Genet 155(2):945-959.

Rosenberg NA 2004 DISTRUCT: A program for the graphical display of population structure. Mol Ecol Notes 4(1):137-138.

Roy MS, Geffen E, Smith D, Ostrander EA, Wayne RK 1994 Patterns of differentiation and hybridization in North American wolflike canids, revealed by analysis of microsatellite loci. Mol Biol Evol 11(4):553-570.

Saitou N, Nei M 1987 The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol Biol 4(4):406-425.

Seo JH, Oh JD, Lee JH, Seo DW, Kong HS 2015 Studies on genetic diversity and phylogenetic relationships of Korean native chicken using the microsatellite marker. Korean J Poult Sci 42(1):15-26.


Suh SW, Sharma A, Lee SH, Cho CH, Kim JH, Choi SB, Kim H, Seong HH, Yeon SH, Kim DH 2014 Genetic diversity and relationships of Korean chicken breeds based on 30 microsatellite markers. Asian-Australasian J Anim Sci 27(10):1399-1405.