서 론
조류인플루엔자 바이러스(avian influenza virus; AIV)는 다양한 야생조류 및 가금류에 감염하는 바이러스로 바이러스 외피막(envelope)에 존재하는 hemagglutinin(HA)과 neuraminidase(NA) 단백질의 항원성에 따라 HA는 16개 아형(subtype), NA는 9개 아형으로 구별된다(Fouchier et al., 2005; Swayne et al., 2013). 닭에 감염하는 AIV는 감염 닭의 75% 이상 폐사를 유발하는 고병원성 AIV(highly pathogenic AIV; HPAIV)와 75% 미만의 폐사를 일으키는 저병원성 AIV(low pathogenic AIV; LPAIV)로 구분된다(OIE, 2012). 16개 HA 아형 AIV 중 H5과 H7 아형 바이러스만 HPAIV 병원성을 발휘할 수 있지만, 대부분의 H5 및 H7 AIV는 LPAIV의 병원성을 보인다(Alexander, 2007).
H5 및 H7형 LPAIV는 여러 기전으로 인하여 HPAIV로 변이될 수 있다(Banks et al., 2001; Duan et al., 2007; Horimoto et al., 1995). 1983년 미국에서 발생한 고병원성 H5N2 바이러스(Kawaoka et al., 1984), 1994년 멕시코에서 발생한 고병원성 H5N2 바이러스(Horimoto et al., 1995), 1999년 이탈리아에서 발생한 고병원성 H7N1 바이러스(Capua et al., 2000), 2003년 네덜란드에서 발생한 고병원성 H7N7 바이러스 (Fouchier et al., 2004) 등은 처음 저병원성이었던 바이러스가 고병원성 바이러스로 변이되어 피해가 유발된 사례들이다.
H7 아형 AIV 중 일부는 사람에 감염하기도 한다. 2004년 네덜란드에서 H7N7 AIV는 사람에 감염하여 대부분은 결막염을 동반하는 가벼운 임상증상을 보였으나, 일부는 사망이 발생하기도 하였다(Fouchier et al., 2004). 2013년부터는 중국에서 H7N9 AIV로 인한 사람 감염이 나타나기 시작하여 많은 피해가 발생되고 있다. H7N9 AIV는 현재(2019년 4월)까지 총 1,568명의 사람 감염이 발생하여 그중 615명이 사망하여 39.2%의 높은 사망률을 보이고 있다(WHO, 2019). 이 바이러스는 사람에게는 높은 병원성을 보이지만, 닭에서는 LPAIV의 병원성을 나타내며, 적어도 3종 이상의 AIV가 유전자 분절의 재편성(reassortment) 과정을 거쳐 신종 바이러스가 출현된 것으로 추정되고 있다(Gao et al., 2013; Lam et al., 2013; Liu et al., 2013).
AIV는 PB2, PB1, PA, HA, NP, NA, M, NS 등 8개 유전자가 분절된 형태로 존재하며, 이들 유전자들의 변이(mutation)나 유전자 분절의 재편성(reassortment) 등으로 새로운 유형의 바이러스가 출현하는 것으로 알려져 있다(Horimoto and Kawaoka, 2001; White and Lowen, 2018).
본 연구는 최근에 야생조류에서 분리되었던 H7 아형 AIV를 대상으로 HA, NA 유전자 등 8개 유전자 전체 염기서열을 분석하여 그 특성과 사람 감염이 있었던 바이러스와 유전자 특성을 비교하고자 실시하였다.
재료 및 방법
바이러스 분리 및 증식은 AIV 항체 음성인 9∼11일령 계태아 종란(성민축산, 한국)을 이용하여 실시하였다. 즉, 채취한 야생조류 분변을 항생제(100 unit/μL of penicillin and 100 mg/μL of streptomycin)가 첨가된 PBS에 혼합하여 충분히 섞어준 후, 혼합물은 3,000 rpm에 10분간 원심분리하여 상층액을 0.45 μm 필터(Millex syringe-driven filters, Millipore, USA)로 여과한 후, 여과액을 9∼11일령 계태아 종란의 요막강(allantoic cavity)내로 접종한 후 37℃에서 4∼5일간 배양하였다. 배양액에서의 AIV 유무 및 아형 확인은 혈구응집반응(OIE, 2012)과 각각의 AIV 아형 특이 primer 및 probe를 이용한 real-time reverse transcription polymerase chain reaction(rRT-PCR)으로 확인하였다(Hoffmann et al., 2016). 바이러스가 분리된 분변의 조류 종 동정(DNA barcoding)은 분변에서 mitochondrial DNA를 추출하여 cytochrome c oxidase I 유전자를 증폭하여 분석하는 방법으로 실시하였다(Lee et al., 2010).
바이러스 염기서열 분석을 위한 RNA 추출 및 증폭 시료는 바이러스를 감염시킨 계태아 요막강액을 사용하여 실시하였다. 즉, AIV 감염 요막강액에서 Viral Gene-spinTM Viral DNA/RNA Extraction Kit(iNtRON Biotechnology, Korea)를 사용하여 RNA를 추출한 후 AIV의 segment 특이 primer를 이용한 reverse transcription polymerase chain reaction(RTPCR) 방법(Hoffmann et al. 2001)으로 각각의 segment 유전자를 증폭하였다. 증폭된 RT-PCR 산물을 QIAquick gel exctraction kit(QIAGEN, USA)로 정제하여 유전자 염기서열 분석 전문회사(Macrogen Inc, Korea)에 의뢰하여 염기서열을 분석하였다. 염기서열 분석 결과는 Molecular evolutionary genetics analysis(MEGA) version 7 software(http://megasoftware.net/)와 MegAlign program(DNASTAR Inc, USA)의 Clustal W Method를 이용하여 분석하였다. 염기서열의 계통유전학적 분석(phylogenetic analysis)은 MEGA 7 software를 이용하여 neighbor-joining method로 1,000 bootstrap replicates로 실시하였다.
결 과
본 연구의 유전자 분석에 사용된 바이러스 분리 내력은 Table 1과 같다. Hoffmann et al.(2016)이 보고한 각각의 아형 특이 primer 및 probe를 사용한 rRT-PCR법으로 분리된 바이러스 아형을 확인한 결과, H7N7이 4주, H7N1이 2주이었다. 바이러스가 분리된 분변을 대상으로 DNA barcoding을 실시하여 분리된 숙주의 종을 동정한 결과, 3주는 청둥오리(Anas platyrhynchos)에서, 3주는 쇠오리(Anas crecca)에서 각각 분리된 것으로 동정되었다.
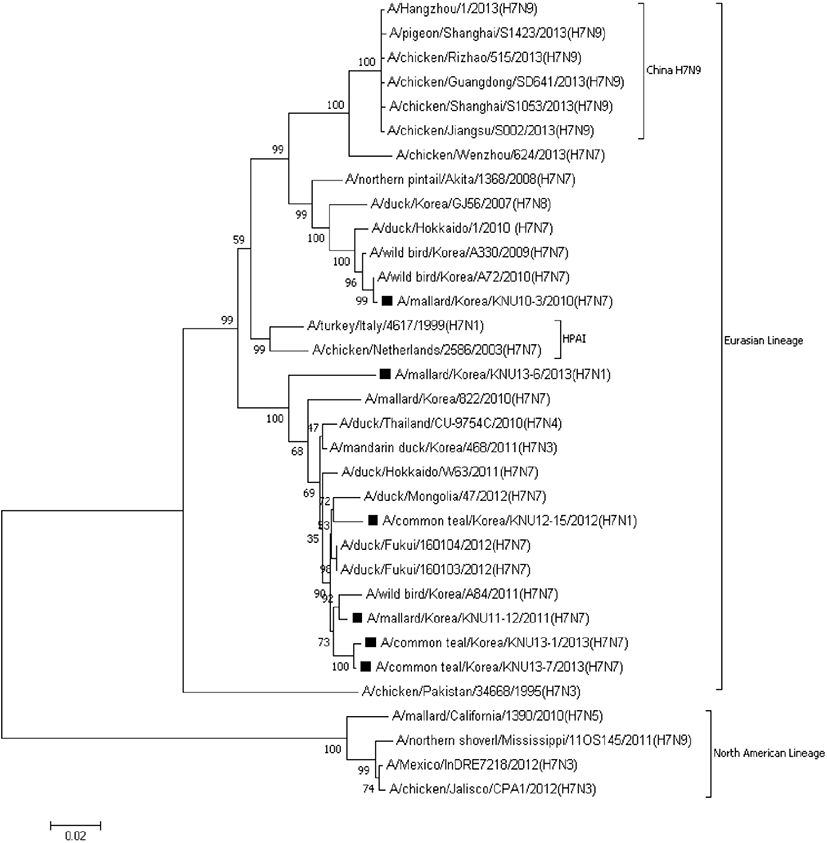
H7 아형의 HA gene을 대상으로 계통유전학적 분석을 실시한 결과, 크게 Eurasian lineage와 North American lineage로 분류되었는데, 한국 야생조류 유래 분리주는 모두 Eurasian lineage로 분류되었다(Fig. 1). Eurasian lineage내에서는 다양한 sublineage로 분류되었는데, 6개 분리주 중 2개 분리주(A/common teal/Korea/KNU13-1/2013, A/common teal/ Korea/KNU13-7/2013)만 같은 sublineage로 분류되었으나 나머지 4개 분리주는 각기 다른 sublineage로 분류되었다. 분리주 중 1주(A/mallard/Korea/KNU10-3/2010(H7N7))는 중국에서 사람 감염이 나타나고 있는 H7N9 sublineage와 가까운 근연 관계를 보였으나, 이들과는 또 다른 별개의 sublineage로 분류되었고, 나머지 5개 분리주들도 중국 사람 감염 H7N9 바이러스와는 다른 sublineage로 분류되었다. 또한, 한국 분리주 모두는 고병원성을 보였던 이탈리아 분리주(A/turkey/Italy/4617/1999(H7N1)와 네덜란드 분리주(A/chicken/Netherlands/2586/2003(H7N7))와는 확연히 다른 sublineage로 분류되었다(Fig. 1).
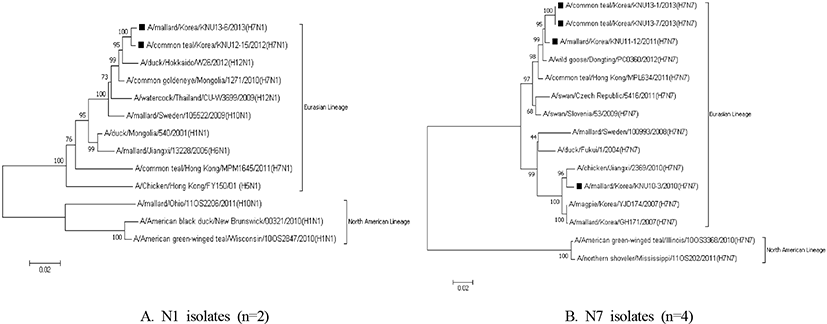
H7N1 아형 2주와 H7N7 아형 4주를 각각 동일한 NA 아형 바이러스들과 계통유전학적 분석을 실시하였다. H7N1 아형으로 분류된 2주의 NA 유전자 계통유전학적 분석결과, 모두 Eurasian lineage로 분류되어 North American lineage와는 확실한 차이를 보였고, 일본에서 분리된 H12N1 아형 바이러스(A/duck/Hokkaido/W26/2012(H12N1))의 NA 유전자와 가장 가까운 근연 관계를 보였다. H7N7 아형으로 분류된 4주도 모두 Eurasian lineage로 분류되었으며, 1개 분리주(A/mallard/Korea/KNU10-3/2010(H7N7))는 중국에서 분리된 H7N7 아형 바이러스(A/chicken/Jiangxi/2369/2010(H7N7))와 가장 가까운 근연 관계를 보였고, 나머지 3주는 중국에서 분리된 H7N7 분리주인 A/wild goose/Dongting/PC0360/2012 (H7N7)와 가장 가까운 근연 관계를 보였다(Fig. 2).
바이러스 내부 유전자인 PB2, PB1, PA, NP, M 및 NS에 대한 유전자 염기서열 분석 결과를 토대로 각각의 유전자에 대한 계통유전학적 분석을 실시하였다. PB2 유전자의 경우, 한국 야생조류 분리주는 모두 Eurasian lineage로 분류되었다. 3개의 분리주(A/common teal/Korea/KNU12-15/2012(H7N1), A/common teal/Korea/KNU13-1/2013(H7N7), A/common teal/ Korea/KNU13-7/2013(H7N7))는 중국 사람 감염 H7N9 바이러스들과 가까운 sublineage에 위치하였으나 별개의 sublineage로 분류되었고 나머지 3주와는 완전히 다른 sublineage로 분류되었다(Fig. 3A).
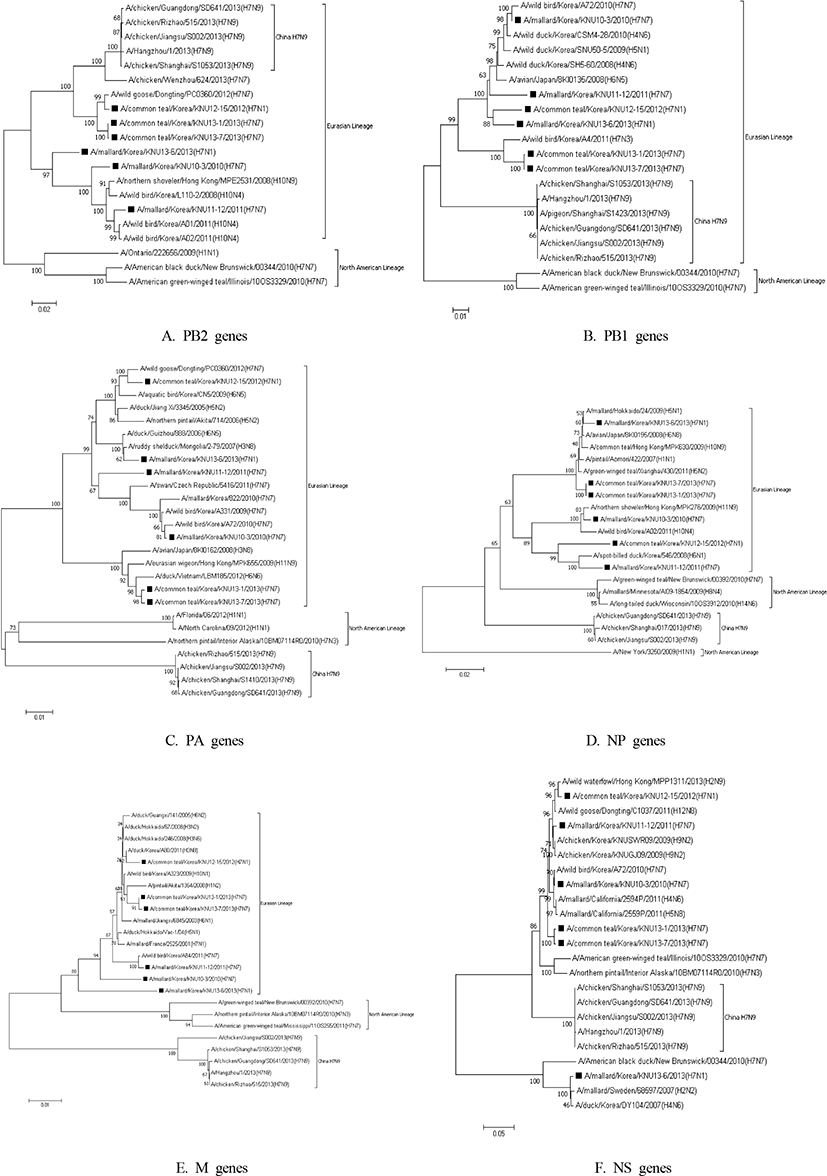
PB1 유전자는 국내 분리주 6주 모두는 Eurasian lineage로 분류되었고, Eurasian lineage 내에서도 적어도 3종 이상의 sublineage로 분류되었다. 중국 사람 감염 H7N9 바이러스들은 Eurasian lineage로 분류되었으나, 국내 분리주들과는 완전히 다른 별개의 sublineage로 분류되었다(Fig. 3B).
PA 유전자는 한국 분리주 6주 모두 Eurasian lineage로 분류되었으며 Eurasian lineage 내에서도 2개 분리주(A/common teal/Korea/KNU13-1/2013(H7N7), A/common teal/Korea/KNU137/2013(H7N7))만 같은 sublineage에 속하였으나, 나머지 4개주는 각기 다른 sublineage로 분류되었다. 중국 사람 감염 H7N9 바이러스의 PA 유전자는 Eurasian lineage에 속하지 않는 것으로 나타나, 국내 분리주 모두는 중국 사람 감염주와는 확연히 다른 계열인 것으로 파악되었다(Fig. 3C).
NP 유전자는 분리주 모두 Eurasian lineage로 분류되었으며, Eurasian lineage 내에서도 다양한 sublineage로 분류되었다. 중국 사람 감염 H7N9 바이러스의 NP 유전자는 Eurasian lineage에 속하지 않는 것으로 나타나, 국내 분리주 모두는 중국 사람 감염주와는 확연히 다른 계열인 것으로 나타났다(Fig. 3D).
M 유전자의 경우, 분리주 모두 Eurasian lineage로 분류되었고, Eurasian lineage 내에서 다양한 sublineage로 분류되었다. 중국 사람 감염 H7N9 바이러스의 M 유전자는 Eurasian lineage로 분류되지 않고, 별개의 lineage로 분류되는 것으로 나타나, 한국 분리주 모두는 중국 분리 사람 감염주와는 완전히 다른 lineage로 분류되었다(Fig. 3E).
NS 유전자의 계통유전학적 분석을 실시한 결과 1개 분리주(A/mallard/Korea/KNU13-3/2013)는 나머지 5개주와 완전히 다른 계열로 분류되었다. 또한, 6개 분리주 모두는 중국 사람 감염 H7N9 바이러스들과는 별개의 계열로 분류되었다(Fig. 3F).
분리된 바이러스의 주요 단백질에 대한 분자 특성을 알아보기 위하여 분석한 염기서열을 토대로 추정 아미노산서열을 비교하여 병원성, 숙주 특이성, 약제 저항성 등 각각의 특성을 비교 분석하였다(Hay et al., 1985; Holsinger et al., 1994; Gubareva et al., 1997; Duan et al., 2007).
HA 단백질 cleavage site의 아미노산을 분석한 결과, 한국 분리주 6주 중 2주(A/common teal/Korea/KNU12-15/2012, A/common teal/Korea/KNU13-1/2013)는 PEIPKGR/G motif를 가지는 것으로 나타났고 나머지 4주는 PELPKGR/G motif를 가지는 것으로 나타나 6주 모두 저병원성 바이러스의 특성을 보였다(Table 2). 모든 분리주는 숙주세포 receptorbinding site(RBS)로 알려진 235번(H3 subtype numbering은 226번) 아미노산은 glutamine(Q), 237번(H3 subtype numbering은 228번)은 glycine(G)을 가지는 것으로 나타나 모두 조류 세포 receptor와 결합 특이성이 높을 것으로 판단된다(Neumann and Kawaoka, 2015). NA 단백질에서 neuraminidase 활성이나 숙주 적응과 관련된 stalk region에서의 결손 여부를 조사한 결과, 한국 분리주 6주 모두는 NA stalk region에서 결손은 관찰되지 않았다(Table 2).
PB2 단백질에서 포유류에서의 감염과 관련된 변이 부위인 E627K 아미노산을 분석한 결과, 한국 분리주 모두는 glutamic acid(E)로 사람 감염 바이러스의 특성을 보이지 않았다. 또한, PB1 단백질에서 포유류 증식과 관련된 변이 부위로 알려진 I368V 아미노산을 분석한 결과, 한국 분리주 모두는 Isoleucine(I)을 가지는 것으로 나타나 변이가 관찰되지 않았다(Table 2).
사람감염 H7N9 바이러스는 NS 단백질의 일정 부위(218∼230) 아미노산 결손이 관찰된다(Chen et al., 2015). 한국 분리주를 대상으로 조사한 결과, 6주 모두 NS 단백질 결손은 관찰되지 않았다. M2 단백질에서 S31N의 변이가 발생하면 바이러스 치료 약제인 amantidine과 rimantadine에 대한 저항성이 생기는 것으로 알려져 있다(Hay et al., 1985). M2단백질의 S31N 변이 여부를 조사한 결과, 한국 분리주 모두는 31번 아미노산이 Serine(S)으로 변이가 나타나지 않았다(Table 2).
고 찰
야생조류에서 분리된 H7 아형 AIV 6주를 대상으로 8개 전체 유전자 염기서열을 분석하고, 그 특성을 조사하였다. 16개 H 아형과 9개 N 아형에 각각 특이적인 primer 및 probe를 사용한 rRT-PCR법(Hoffmann et al., 2016)으로 분리된 바이러스의 아형을 확인한 결과, H7N7 아형 4주, H7N1 아형 2주로 분류되었다. 2005∼2014년간 국내에서 분리된 H7형 분리주 29주를 대상으로 아형을 조사한 결과, H7N7 아형이 65.5%(19/29)로 가장 흔하게 분리된 것으로 보고된 바 있는데, 본 연구에서도 다수가 H7N7 아형으로 나타나 이들의 보고와 비슷하였다(Kim et al., 2016).
8개 유전자 전체 염기서열을 분석하여 국내 및 외국 분리주들과의 계통유전학적 근연 관계를 분석하였다. H7 아형 바이러스의 표면 유전자인 HA와 NA 유전자를 대상으로 계통유전학적 분석을 실시할 경우, 크게 몇 가지의 lineage 즉, Eurasian, Australian 및 North American lineage 등으로 분류된다(Bulach et al., 2010; Kang et al., 2014). 본 실험에서 분리된 6주의 HA 유전자를 대상으로 phylogenetic tree를 작성한 결과, 국내 분리주 6주 모두는 Eurasian lineage로 분류되었다. Eurasian lineage 내에서도 국내분리주들은 2주를 제외하고는 각기 다양한 sublineage로 분류되어 유전적 다양성이 관찰되었다. HA 유전자는 중국에서 사람 감염이 나타나고 있는 H7N9 바이러스 sublineage와는 가까운 근연 관계를 보이는 바이러스도 있었지만, 6주 모두 중국 사람 감염 바이러스와는 별개의 sublineage로 분류되었다. 국내 야생조류에서 분리된 H7 아형 AIV에 대한 다른 연구자들의 계통유전학적 분석 결과에서도 국내 야생조류 분리주들은 Eurasian lineage에 속하며, Eurasian lineage 내에서 다양한 sublineage로 재분류되는 것으로 확인된 바 있었는데, 본 연구에서도 이들의 연구결과와 유사한 양상을 보였다(Kang et al., 2014; Kim et al., 2016).
바이러스 내부 유전자의 계통유전학적 분석을 실시한 결과, PB2 유전자의 경우 야생조류 유래 국내 분리주는 모두 Eurasian lineage로 분류되었다. 그러나 Eurasian lineage 내에서도 H7N1 분리주 1개(A/common teal/Korea/KNU12-15/ 2012(H7N1))와 H7N7 분리주 2주(A/common teal/Korea/ KNU13-1/2013(H7N7), A/common teal/Korea/KNU13-7/2013 (H7N7))는 나머지 3주와는 완전히 다른 sublineage로 분류되었다. 또한, 별개의 sublineage로 분류된 3주도 각기 다른 sublineage 형태로 분류되어 PB2 유전자 역시 유전적 다양성이 있는 것으로 판단된다. 국내 분리주에 대한 PB2 유전자 분석결과, 국내 분리주는 적어도 3종 이상의 clade를 구성하는 것으로 분석된 바 있는데, 본 연구 결과에서도 PB2 유전자도 다양성이 있는 것으로 나타나 이들 연구결과와 비슷한 결과를 보였다(Kang et al., 2014; Kim et al., 2016).
내부 유전자중 PA, NP 및 M 유전자 계통유전학적 분석을 실시한 결과 국내 분리주들은 모두 Eurasian lineage에 속하였으나, Eurasian lineage 내에서 다양한 sublineage로 분류되는 것으로 나타났다. 중국 사람 감염 H7N9 바이러스는 이들 3개 유전자 모두 Eurasian lineage와는 다른 lineage로 분류되는 것으로 나타났다. 따라서 국내 분리주들은 PA, NP 및 M 유전자는 중국 사람 감염 H7N9 바이러스와는 많은 차이가 있을 것으로 판단된다.
중국 사람 감염 H7N9 바이러스는 적어도 4개 이상의 기원(origin)을 가지는 다양한 reassortment로 인하여 출현한 신종 바이러스로 알려져 있다. 즉, HA 유전자는 사육 오리에 감염하는 AIV에서 유래되었고, NA 유전자는 야생조류 감염 AIV로부터 유래되었으며, 내부 유전자는 가금류에 감염하는 적어도 2종 이상의 H9N2 바이러스로부터 유래된 것으로 알려져 있다(Kageyama et al., 2013; Liu et al., 2013). 본 실험에서 분리된 바이러스의 내부 유전자를 분석한 결과, 중국 사람 감염주와 같은 sublineage로 분류되는 바이러스가 없었으며 특히, PA, NP 및 M 유전자는 중국 사람 감염 H7N9 바이러스와는 별개의 lineage로 구성되는 것으로 보아, 가금류의 H9N2 바이러스 유래 내부 유전자를 가지는 중국의 H7N9 바이러스와는 내부 유전자가 완전히 다른 것으로 판단된다.
AIV 병원성은 HA 단백질의 cleavage site가 염기성 아미노산인 arginine(R)과 lysine(K)을 다수 가지는 형태 즉, HA1 단백질의 C 말단 4개 아미노산중 3개 이상이 염기성 아미노산일 경우 HPAIV로 분류된다(Senne et al., 1996; Horimoto & Kawaoka, 2001). 국내 분리주의 HA 단백질 cleavage site의 아미노산을 분석한 결과, 6주 중 2주는 PEIPKGR/G motif, 나머지 4주는 PELPKGR/G motif를 가지는 것으로 나타났다. 따라서 HA1 단백질 C 말단 4개의 아미노산에서 각각 2개의 염기성 아미노산을 가지는 것으로 나타나 국내 야생조류 유래 분리주 모두는 LPAIV로 판단된다. H7형 AIV 중 본 실험에서 분석한 H7N1 및 H7N7 바이러스와 동일한 아형 중에서 저병원성 바이러스가 고병원성 AIV로 변이된 경우가 1999년 이탈리아와 2003년 네덜란드에서 각각 보고된 바 있다(Capua et al., 2000; Fouchier et al., 2004). 따라서 현재 야생조류에서 분리되고 있는 H7N1과 H7N7 바이러스가 가금류에 전파하여 유행할 경우 병원성 변이 가능성도 배제할 수 없기 때문에 가금류로의 전파가 되지 않도록 하는 방역조치가 필요할 것으로 생각된다.
바이러스 단백질중 감염 숙주 특이성과 관련이 많은 것은 HA 단백질의 receptor-binding site(RBS)로 이중 235번(H3 subtype numbering은 226번)과 237번의 아미노산이 각각 leucine(L)과 serine(S)으로 변이될 경우 사람 감염 가능성이 높아지는 것으로 알려져 있다. 본 실험의 국내 분리주 모두 235번 아미노산은 glutamine(Q), 237번 아미노산은 glycine(G)인 것으로 나타나 조류 세포의 감염 특이성이 유지되고 있는 것으로 판단된다.
NA 단백질중 stalk region의 아미노산 결손으로 초래되는 stalk region의 길이 변화는 influenza virus의 병원성에 영향을 미치며, 포유류 감염 가능성을 증가시키는 것으로 알려져 있다(Zhou et al., 2009; Munier et al., 2010; Neumann and Kawaoka, 2015). 중국 사람 감염 H7N9 바이러스는 NA stalk region의 5개 아미노산(69∼73) 결손이 관찰된다(Chen et al. 2015). 국내 야생조류 분리주들의 NA stalk region에서 결손 여부를 조사한 결과, 분리주 모두는 NA stalk region의 결손은 나타나지 않아 중국 사람 감염주와는 차이를 보였다.
PB2 단백질의 627번 아미노산이 glutamic acid(E)가 lysine(K)으로 변이될 경우와 PB1 단백질의 368번 아미노산 Isoleucine(I)이 valine(V)으로 변이될 경우, 사람 감염 가능성이 높아지는 것으로 알려져 있다(Neumann and Kawaoka, 2015). 국내 분리주는 PB2의 627번 아미노산과 PB1의 368번 아미노산이 각각 glutamic acid(E)와 Isoleucine(I)으로 변이가 관찰되지 않아 사람 감염 가능성은 낮을 것으로 판단된다.
M2 단백질에서 S31N의 변이가 발생하면 바이러스 치료 약제인 amantidine과 rimantadine에 대한 내성이 생기는 것으로 알려져 있다(Hay et al., 1985). M2 단백질의 S31N 변이 여부를 조사한 결과, 한국 분리주 모두 31번 아미노산이 Serine(S)으로 변이가 나타나지 않아 이들 약제에 대한 내성이 생기지 않은 것으로 판단된다.
이상의 결과들을 종합하면 국내 야생 조류에서 분리된 H7 아형 바이러스 6주 모두는 HA 등 바이러스 표면 유전자와 내부 유전자들 모두가 최근 중국에서 사람 감염이 나타나는 H7N9 바이러스와는 차이가 있고, 사람 감염 가능성이 있는 유전자 변이도 관찰되지 않는 것으로 보아 국내 분리 H7 아형 바이러스들은 사람 감염 가능성이 낮을 것으로 판단된다.
국내 야생조류에서 분리된 6주 모두는 가금류에서 저병원성을 나타내는 분자 특성을 보이는 것으로 나타났다. 그러나 국내 분리주와 동일한 H7N1 아형과 H7N9 아형 바이러스는 처음에는 저병원성이었으나, 가금류에 감염하여 HPAIV로 변하는 경우도 있기 때문에 야생조류로부터 가금류로의 전파가 이루어지지 않도록 철저한 방역조치가 필요하고, 야생조류에서의 지속적인 모니터링이 필요할 것으로 판단된다.
적 요
조류인플루엔자 바이러스 H7 subtype에 속하는 바이러스중 일부는 가금류에 감염할 경우 고병원성이 발휘된다. 또 H7 아형 AIV중 일부는 사람에 감염하여 사망 등을 유발할 수도 있다. 본 연구는 야생조류로부터 분리된 H7 아형 조류인플루엔자 바이러스 6주(H7N7 아형 4주, H7N1 아형 2주)를 대상으로 8개 유전자 분절 전체의 염기서열을 분석하여 병원성, 사람 감염 가능성 등 그 특성을 조사하였다. 계통유전학적 분석결과, 국내에서 분리된 H7 아형 분리주들은 8개 유전자(HA, NA, PB2, PB1, PA, NP, M, NS) 모두 Eurasian lineage로 분류되었으나, Eurasian lineage 내에서도 각기 다른 sublineage로 분류되어 유전적 다양성이 있는 것으로 분석되었다. 한국 분리주 6주는 HA 단백질 분절부위 아미노산은 두 종류(PEIPKGR 및 PELPKGR)의 motif를 가지고 있었으나, 모두 저병원성 바이러스 특성을 가지고 있었다. 숙주세포 결합 특이성과 관련 있는 HA 단백질 receptor-binding site를 분석한 결과, 한국 분리주 모두는 사람 세포 수용체 결합 특이성보다는 조류 세포 수용체 결합 특이성을 가지는 것으로 나타났다. 사람 감염 가능성을 높게 하는 부위에서의 아미노산 치환(PB2 단백질의 E627K 및 PB1단백질의 I368V)도 나타나지 않았고, 또한 NA stalk region에서의 결손도 관찰되지 않았다. 이상의 결과를 미루어 볼 때 한국 야생조류에서 분리된 H7 아형 6주 모두는 저병원성 바이러스로 최근 중국에서 사람 감염이 나타나고 있는 H7N9 바이러스와는 유전적으로 다른 계열의 바이러스인 것으로 판단된다.
(색인어: 조류인플루엔자 바이러스, H7 아형, 유전자, 계통유전학적 분석, 야생조류)








