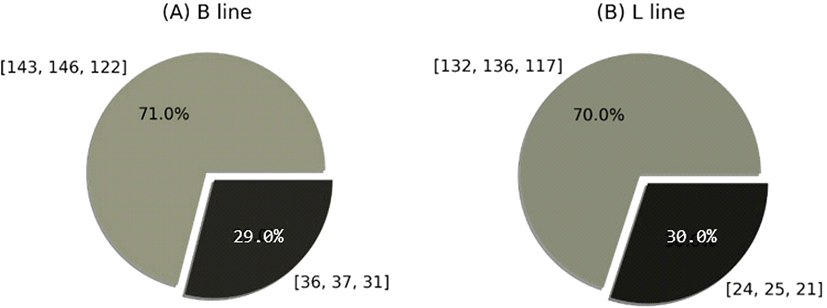Article
K-Means Clustering으로 분류한 닭 깃털색 표현형의 분석
박종호1
,
허선영1
,
김민준2
,
조은진1
,
차지혜3
,
진대혁4
,
고영준5
,
이승환5,6
,
이준헌5,6,†
Analysis of Chicken Feather Color Phenotypes Classified by K-Means Clustering using Reciprocal F2 Chicken Populations
Jongho Park1
,
Seonyeong Heo1
,
Minjun Kim2
,
Eunjin Cho1
,
Jihye Cha3
,
Daehyeok Jin4
,
Yeong Jun Koh5
,
Seung-Hwan Lee5,6
,
Jun Heon Lee5,6,†
Author Information & Copyright ▼
1Graduated Student, Department of Bio-AI Convergence, Chungnam National University, Daejeon 34134, Republic of Korea
2Graduated Student, Division of Animal and Dairy Science, Chungnam National University, Daejeon 34134, Republic of Korea
3Researcher, Animal Genome & Bioinformatics, National Institute of Animal Science, Rural Development Administration, Wanju 55365, Republic of Korea
4Researcher, Animal Genetic Resources Research Center, National Institute of Animal Science, Rural Development Administration, Hamyang 50000, Republic of Korea
5Professor, Department of Bio-AI Convergence, Chungnam National University, Daejeon 34134, Republic of Korea
6Professor, Division of Animal and Dairy Science, Chungnam National University, Daejeon 34134, Republic of Korea
© Copyright 2022, Korean Society of Poultry Science. This is an Open-Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Sep 05, 2022; Revised: Sep 18, 2022; Accepted: Sep 19, 2022
Published Online: Sep 30, 2022
적요
RGB 조건에서 하나의 픽셀은 255의 세제곱 개 이상의 색상을 표현할 수 있다. 현재까지의 컴퓨터 비전 연구는 조류에서 나타나는 다양한 깃털색 표현형에 대해 세밀히 분석하여 종을 구분하였지만, GWAS에 이용될 목적을 위해 다양하게 유전되는 색상을 단순화하지 못하였다고 판단된다. 본 연구는 연산오계(YO)와 백색레그혼(WL) 상호 역교배 F2 집단을 이용하였으며 이미지 양자화를 통하여 이미지의 크기를 줄이고 저장을 용이하게 하였으며 깃털색의 원인 유전자 탐색을 위한 기초 자료를 제공하기 위하여 육안으로 결정하였던 다양한 깃털색을 단순화하였다. 특히, GWAS 연구에 필요한 수치화된 표현형을 제시하였다는 측면에서 가치가 있다고 판단된다.
ABSTRACT
Chickens are a species of vertebrate with varying colors. Various colors of chickens must be classified to find color-related genes. In the past, color scoring was performed based on human visual observation. Therefore, chicken colors have not been measured with precise standards. In order to solve this problem, a computer vision approach was used in this study. Image quantization based on k-means clustering for all pixels of RGB values can objectively distinguish inherited colors that are expressed in various ways. This study was also conducted to determine whether plumage color differences exist in the reciprocal cross lines between two breeds: black Yeonsan Ogye (YO) and White Leghorn (WL). Line B is a crossbred line between YO males and WL females while Line L is a reciprocal crossbred line between WL males and YO females. One male and ten females were selected for each F1 line, and full-sib mating was conducted to generate 883 F2 birds. The results indicate that the distribution of light and dark colors of k-means clustering converged to 7:3. Additionally, the color of Line B was lighter than that of Line L (P<0.01). This study suggests that the genes underlying plumage colors can be identified using quantification values from the computer vision approach described in this study.
Keywords: chicken; image quantization; computer vision; k-means clustering; plumage color
서 론
GWAS(genome-wide association study)는 유전체에 존재하는 많은 SNP(single nucleotide polymorphism)들을 이용하여 유전체 전체를 대상으로 원인이 되는 가장 유의성이 있는 SNP를 찾는 데 사용하는 방법이다(Korte and Farlow, 2013). 이는 인간에서 질병에 대한 원인 변이를 찾기 위하여 많이 연구되어 왔으며 가축의 경우 질병뿐 아니라 경제형질에 대한 연구도 많이 이루어져왔다. 또한 최근에는 피모색과 같이 색과 관련된 후보 유전자 변이를 찾기 위하여 GWAS가 이용되기도 하였다(Nie et al., 2016; Luo et al., 2018; Mastrangelo et al., 2020). 연관 분석은 원인이 되는 효과를 가진 유전 변이체를 식별하는 기술로써 유의한 결과를 도출하기 위하여 분석에 이용되는 표현형이 정확할수록 결과의 도출이 쉽다고 일반적으로 알려져 왔다(Banerjee et al., 2018). 따라서 정확한 표현형의 측정은 실험의 결과에 많은 영향을 미치고 있으며 많은 연구자들이 표현형의 정확도 향상에 노력을 들이고 있다. 특히 색깔과 같이 사람이 판단하여 분류하는 경우 보는 각도, 빛의 유무, 눈의 상태에 따라서 기준이 달라질 수 있기 때문에 정확한 분류의 기준을 설정할 필요가 있다.
최근에 인간의 주관적인 기준을 대체하기 위해 컴퓨터 비전을 많이 사용하고 있다. 컴퓨터 비전에서 데이터 단위는 가시 스펙트럼의 색상을 나타내는 이미지 픽셀로 구분이 되는데 픽셀은 픽셀 근접성, 방향 및 유사성이 그룹으로 배열이 되게 된다. 컴퓨터 비전은 이런 픽셀 값을 사용하여 이미지의 내용을 파악하는 컴퓨터 과학의 한 형태이다(LeCun et al., 2015). 컴퓨터 비전을 이용하여 생물다양성을 관찰하면, 비용과 노동력을 줄일 뿐만 아니라 표현형 관찰시 객관성을 부여할 수 있다. 예를 들어, 동물에서 돌고래나 쥐가오리에서 외형의 패턴값을 파악하고 인식하기 위해서 컴퓨터 비전을 이용하였으며(Bouma et al., 2018; Moskvyak et al., 2019), 식물 분야에서도 식물 기관들의 형태적 변화의 측정에 컴퓨터 비전을 활용하여 식물의 성장과 발달을 파악하고 있다(Li et al., 2020).
닭에서 나타나는 여러가지 색과 패턴을 객관화하기 위해서 이미지 양자화를 사용할 수 있다. 컬러 이미지에서 보여지는 모든 픽셀은 Red, Green, Blue 값의 결과로써 이 세 색깔의 다양한 비율을 혼합하여 광범위한 색상을 얻을 수 있다. 이미지 양자화는 이런 다양한 색상을 단순화하고 줄이는 기술이다. 일반적으로 이 기술은 이미지를 압축하고 분할하는 간단한 전처리 단계에서 많이 사용되어 왔다(Yang and Tsai, 1998; Deng et al., 2001). 더 나아가 이미지를 효율적으로 인덱싱하여 대규모 데이터베이스에서 유사성을 검색하기 쉽게 할 뿐만 아니라 새로운 색상 질감 분석 방법에 이용되기도 하였다(Deng et al., 2001; Sertel et al., 2009).
이미지 양자화를 위해 이용되는 기법에는 분할 알고리즘과 클러스터링 기반 알고리즘이 있다. 분할 알고리즘은 원본 이미지의 색 공간을 원하는 색상 수를 얻을 때까지 반복 프로세스를 적용하는 것이며, 클러스터링 기반 알고리즘은 사전에 색상 수를 결정한 다음, 반복적으로 개선하는 것이다. 일반적으로 이미지 양자화를 위해 두 알고리즘을 이용할 수 있는데 클러스터링 기반 알고리즘은 분할 알고리즘보다 계산 시간이 더 걸리는 대신 더 높은 품질의 결과를 얻을 수 있다고 알려져 있다(Ozturk et al., 2014).
색상 양자화에 주로 이용되는 클러스터링 알고리즘에는 k-means(Kasuga et al., 2000; Celebi, 2011), 경쟁학습(Scheunders, 1997), 퍼지 c-means(Özdemir and Akarun, 2002), 개미 알고리즘(Pérez-Delgado, 2015)이 있다. k-means 알고리즘은 가장 보편적으로 사용되는 방법이지만, k를 정해야 한다는 단점이 존재한다. 경쟁학습과 퍼지 c-means는 k-means의 k값의 영향에 더 둔감한 특징이 있지만, 계산 부하가 높아진다는 단점이 존재한다. 한편, 개미 알고리즘은 가장 오류가 적은 방법이면서 클러스터의 수를 알 필요가 없지만, 시간이 많이 걸릴 수 있다는 단점이 있다.
따라서 본 연구의 목적은 기존 인간에 의해 분류한 주관적 평가의 한계를 해결하기 위해 이미지 양자화 기술을 이용하여 색상 및 패턴을 수치화하고, 가장 일반적이고 대중적인 방법이면서 계산이 빠른 k-means clustering을 이용하여 다양한 닭 깃털색 표현형을 분류하고자 하였다.
재료 및 방법
1. Animals
본 연구에서는 연산오계(Yeonsan Ogye: YO)와 화이트레그혼(White Leghorn: WL)을 cross-breeding하여 생산된 F2 generation 집단을 사용하였다. YO는 국내 재래닭 중 유일한 가금 천연기념물로, 피부, 근육 및 골격이 검다는 특징을 가지고 있으며(Lee, 2019) 이와 대조적으로, WL은 전세계적으로 가장 널리 알려진 산란계 품종으로 백색 피모색을 가지고 백색란을 낳는 특징이 있다.
본 연구에 사용된 집단은 부모세대(F0) 수컷 1수와 암컷 5수로 이루어진 2개의 라인(B라인, L라인)으로 구성되어 있다. F1 세대는 F0의 암컷 5수에 각각 1개의 라인으로 총 5개의 라인으로 구성되어 있고, F2 집단을 구성하기 위하여 F1 각 라인 별로 수컷 1수와 암컷 10수를 선발하여 전형매 교배를 실시하였다. 최종적으로 생산된 F2 세대를 본 분석에 활용하였으며, B라인은 F0의 수컷이 YO, 암컷이 WL이고, L라인은 F0의 수컷이 WL이고 암컷이 YO이다. 사용된 F2는 B라인이 468수, L라인이 415수다. 시험에 관련된 닭의 관리 및 취급은 충남대학교 동물실험윤리위원회(IACUC, No. 202103A-CNU-061) 승인을 얻은 후 본 규정을 준수하여 시행하였다.
2. Image Collection
표현형을 분류하기 위한 RGB(Red, Green, Blue) 값을 생산하였으며 그 수집하는 과정은 다음과 같다. 집단 내 개체 별 깃털색 표현형을 조사하기 위해서 (1) 날개를 인위적으로 펴게 했을 때와 (2) 날개를 접었을 때, 총 2장의 사진을 촬영하였다. 본 분석에서는 보다 정확한 표현형을 나타내는 날개를 인위적으로 들고 찍은 사진 데이터만을 사용하였다. 모든 사진은 카메라(D80, Nikon, Tokyo, Japan)를 사용하여 배경, 조명, 각도, 개체 방향을 동일하게 세팅한 환경에서 촬영하였다.
각각의 개체는 배경과 개체의 구분을 정확하게 하기 위해 파이썬(Python version 3.8.0; Python Software Foundation, 2019) 환경에서 배경을 제거하였다. 본 연구에서 필요한 깃털색에 해당하는 정보를 추출하기 위해 Adobe Photoshop CC 2021(Adobe, Mountain View, CA, USA)을 이용하여 개체의 머리와 몸통, 다리를 분리하였다. 머리와 몸통, 몸통과 다리 부분 중, 색깔이 확연히 달라지는 지점을 기준으로 이미지를 분리하였으며, 이후 수행되는 분석에서는 몸통 부분에 해당하는 이미지를 사용하였다.
3. Average RGB Values and Standardization
이미지들은 RGB 색상 조건에서 PNG-24비트 형식으로 처리되었다. 개체의 색상 및 패턴을 수치화하기 위해 해당 이미지 픽셀의 R값, G값, B값의 개체별 평균 및 표준편차를 구하였다. 수치화된 값은 라인별 차이를 확인하기 위해 PCA와 k-means clustering에 사용되었다.
4. K-Means Clustering
닭의 피모색에 해당하는 픽셀들을 모두 모아 RGB 각각에 대해서 k-means clustering을 진행하였다. 그 후 모든 라인의 픽셀 값들 중 반을 무작위 선발하였다. 사용된 집단 내 깃털의 대표 색이 검은색, 하얀색이기 때문에, k는 2로 설정하였으며 k-means clustering은 파이썬 환경에서 scikit-learn 1.0.2에 KMeans 모듈을 사용하였다.
5. Image Quantization
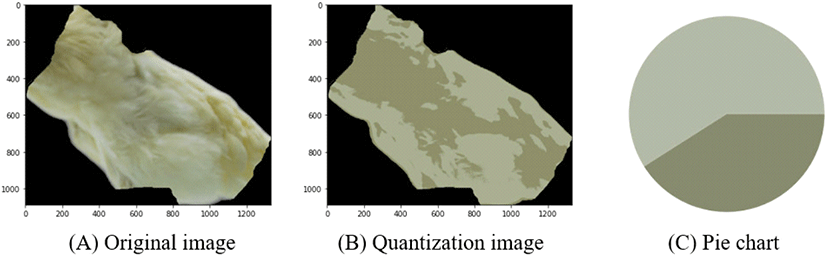
K-means clustering을 할 때, 이미지의 픽셀 수에 R, G, B 개수인 3을 곱해 이미지를 재배열한다. Clustering 이후, centroid 값을 모든 픽셀들에 적용시키면, 이미지는 특정 색상의 수만을 가지게 된다. 본 연구에서는 B와 L라인의 t-test의 결과를 확인하기 위하여 이미지 양자화를 진행하였으며 이미지 양자화한 사진은 Fig. 1에서 제시된 바와 같다.
Fig. 1.
The image quantization and pie chart using quantization image. (A) Original image, (B) quantization image, (C) pie chart using quantization image.
Download Original Figure
결 과
1. PCA와 t-test
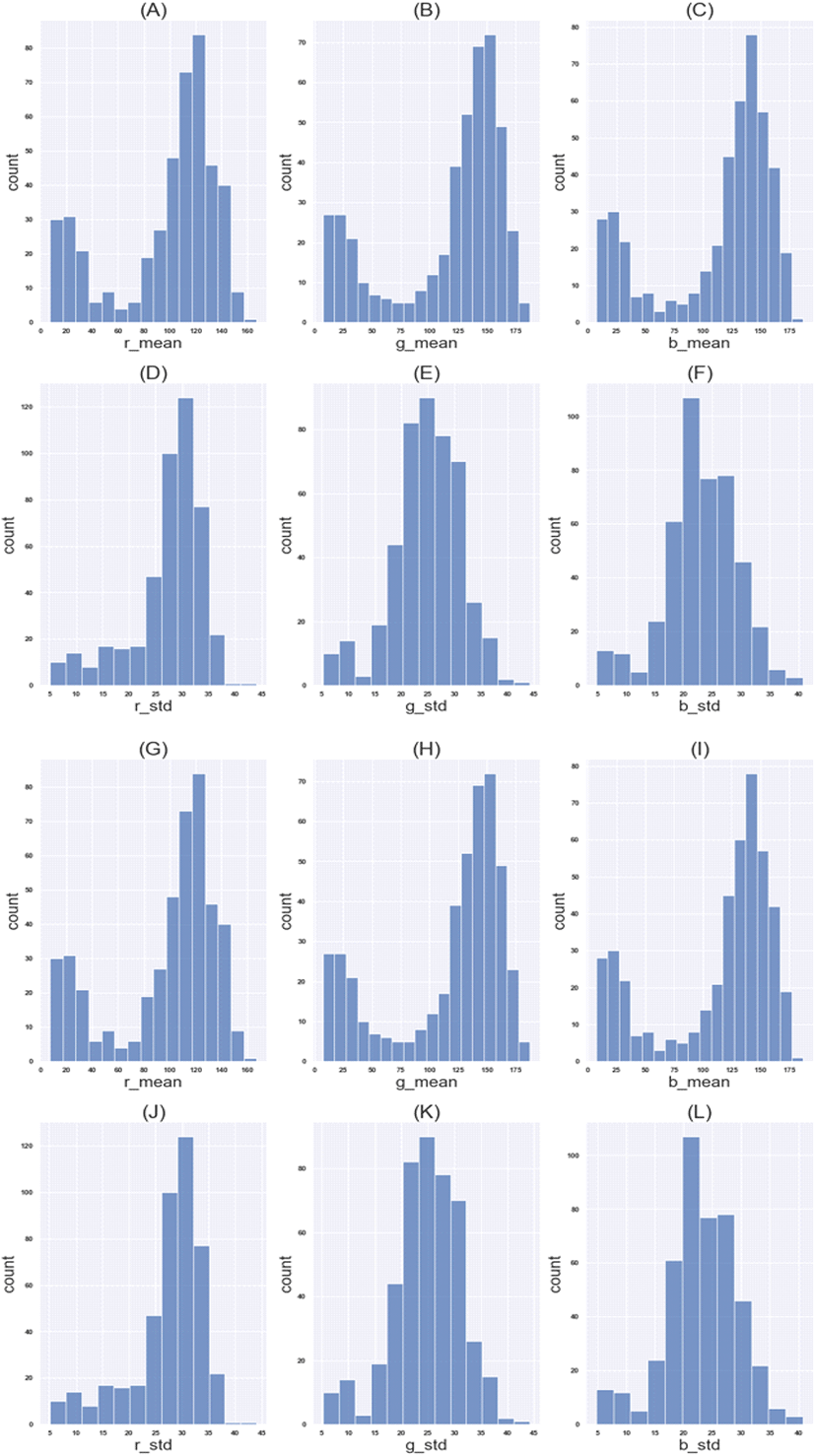
Fig. 2는 한 개체에서 얻어진 B와 L 라인의 평균과 표준편차에 대한 히스토그램 결과이다. Fig. 2에서 확인된 바와 같이 평균은 B와 L 라인 모두 중간값을 나타내는 개체는 빈도가 적으며, RGB 평균값이 낮은 부분과 높은 부분에서 중간값에 비해 상대적으로 높은 빈도를 보이는 것을 확인할 수 있었다. 또한 표준편차는 Fig. 2에서 나타난 바와 같이 B와 L 라인 모두 종모양에 가까운 모습을 확인할 수 있다. 지나치게 단색인 개체와 깃털색이 두개의 분리된 색을 가진 표현형의 개체수는 적었으며, 상대적으로 적당하게 두 개의 깃털색상이 섞여 있는 개체수가 많았다.
Fig. 2.
The distributions of average and standard deviation of RGB values using F2 individual in line B (A)∼(F). (A)∼(C) Histogram for means and their distribution of R, G and B values respectively. (D)∼(F) Histogram for standard deviation and their distribution of each R, G and B values respectively. The distributions of average and standard deviation of RGB values using F2 individual in line L (G)∼(L). (G)∼(I) Histogram for means and their distribution of R, G and B values respectively. (J)∼(L) Histogram for standard deviation and their distribution of R, G and B values respectively.
Download Original Figure
두 라인의 분포의 차이를 확인하기 위해 라인 별로 t-test를 수행하였다. t-test를 수행한 결과, B, L 라인간의 RGB 평균값 모두에 대해서 모두 유의적인 결과를 확인하였다(p-value<0.01)(Table 1).
Table 1.
The t-test for mean values for R, G and B values in on B and L lines
| Line |
P-value |
| |
B |
L |
| Mean of R values |
95.34 |
85.50 |
0.001 |
| Mean of G values |
113.47 |
98.95 |
<0.001 |
| Mean of B values |
111.69 |
96.85 |
<0.001 |
Download Excel Table
2. Average Values of RGB
두 라인간에 유의한 차이를 숫자로 확인하기 위해 k-means clustering을 기반으로 이미지 양자화를 진행하였다. B와 L라인 각각 전체의 픽셀을 추출해 똑같이 k값을 2로 두고 k-means clustering을 실시하여 파이 차트로 나타낸 결과는 Fig. 3과 같다. B라인의 2가지 대표 색상의 RGB 값은 (143, 148, 122), (36, 37, 31)로 확인되었으며 RGB 값이 높은 것을 밝은 부분, RGB 값이 낮은 것을 어두운 부분이라고 표현하였으며 그 비율은 각각 71%, 29% 이었다. L 라인의 대표 RGB 값은 밝은 부분이 (132, 136, 117), 어두운 부분이 (24, 25, 21)로 나뉘었고, 비율은 각각 70%, 30%로 확인되었다.
Fig. 3.
Image quantization results for B (A) and L (B) lines using k=2. The RGB values of the two representative colors of B Line are (143, 148, 122), (36, 37, 31), and their ratios are 71% and 29%, respectively. The RGB values of the two representative colors of L line is (132, 136, 117), (24, 25, 21), respectively, and their ratios are 70% and 30%, respectively.
Download Original Figure
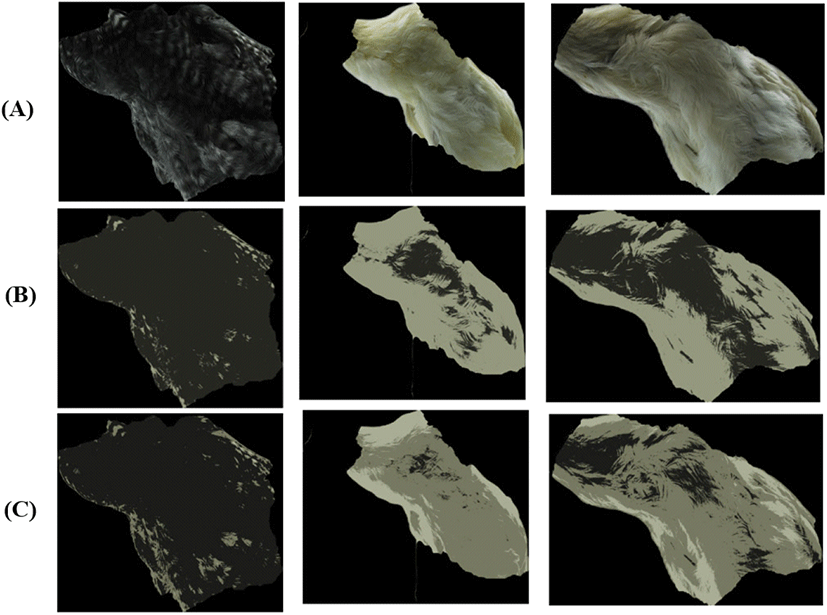
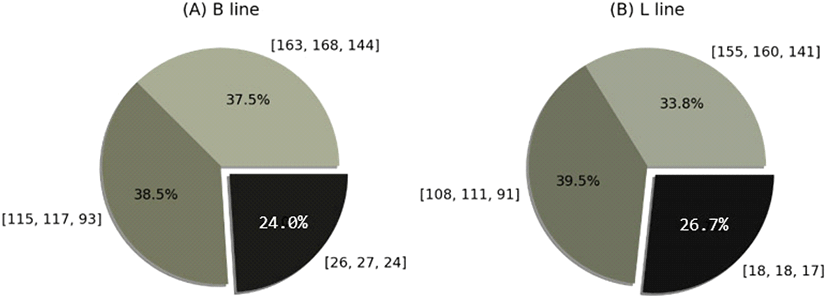
두 라인 간의 이미지 양자화를 위하여 k = 2로 설정을 하였으나 k = 2가 맞는 결과를 도출하는지 확인하기 위하여 k = 3를 이용하여 이미지 양자화를 진행하였다. 이미지 양자화를 통해 나타난 닭의 이미지는 Fig. 4에 제시된 바와 같으며 라인 내에서 이미지를 이용하여 파이 차트를 나타낸 결과는 Fig. 5에 나타난 바와 같다. 색 분포의 비율은 B라인에서 밝은 순서대로 37.5%, 38.5%, 24%가 확인되었고, 각각의 대표 RGB 값은 (163, 168, 144), (115, 117, 93), (26, 27, 24)로 확인되었다. 마찬가지로 L라인은 33.8%, 39.5%, 26.7%가 확인되었고, 각각의 대표 RGB 값은 (155, 160, 141), (108, 111, 91), (18, 18, 17)가 도출되었다. 이 경우 RGB 값이 모두 100 이상이면, 밝은 색이라고 할 수 있다.
Fig. 4.
The example of image quantization results using k=2 and k=3 in B line. The original picture of an F2 bird (A), the image quantization result using k=2 (B), the image quantization result using k=3 (C).
Download Original Figure
Fig. 5.
Image quantization results for B (A) and L (B) lines using k=3. The RGB values of the three representative colors of Line B are (163, 168, 144), (115, 117, 93), (26, 27, 24), and their ratios are 37.5%, 38.5%, and 24%, respectively. The RGB values of the three representative colors of Line L are (155, 160, 141), (108, 111, 91), (18, 18, 17), and their ratios are 33.8%, 39.5%, and 26.7%, respectively.
Download Original Figure
k가 2일 때와 마찬가지로 k가 3일 때, B라인이 L라인에 비하여 조금 더 밝았고, 두 분석결과 모두 나뉘어진 색상 밝기의 차이는 분명했다. 밝은 색을 가진 분포들(대표 RGB 색상 값이 100 이상)에서 각각 (8, 8, 3), (7, 6, 2)의 차이가 확인되었고, 어두운 것에서 (8, 9, 7)의 차이가 확인되었다(Fig. 5). 두 라인을 밝은 부분과 어두운 부분으로 나뉘었을 때, B 라인은 76%와 24%로 나뉘었고 L라인은 73.3%와 26.7%로 나뉘었다. 즉 k를 3으로 하였을 때 또한 분포는 비슷하게 나왔지만, B와 L라인 3가지 각각의 대표색상에 대해서 B라인의 RGB 값이 L라인의 RGB 값보다 약 10정도 밝은 수치를 보였다.
고 찰
1. Distribution of Colors
본 연구에서 이용된 상호 교배 집단, B와 L 라인이 밝은 부분과 검은색 부분의 비율이 7:3인 것을 확인할 수 있었다(Fig. 3). 닭은 8,000여년 전에 인간에 의해 길들여 졌다고 알려져 있으며(Fumihito et al., 1996), 화이트 레그혼(WL)은 지난 100년간 계란 생산을 위한 산란계로 개량되어 왔다. 이 과정에서 야계(wild jungle fowl)와 WL 사이에 많은 차이가 생겼으며 WL은 야생 닭과 비교하여, 특히 계란의 생산이라는 측면에서 야계에 비하여 월등히 높은 성적을 나타내었다(Kerje et al., 2003; Kerje et al., 2004). WL은 대표적인 산란계로서 고정된 상태로 선발되어 한국에 기원한 오계보다 유전적 다양성이 적고, 산란관련 경제형질과 연관된 유전자형을 많이 가지고 있기 때문이라고 추측할 수 있다.
k = 3으로 나타낸 이미지 양자화 결과에서도 Fig. 3의 B, L라인의 분포와 크게 다르지 않은 것을 확인하였다(Fig. 5). 그리고 k = 3에서 나뉘어진 세 부분을 하얀색, 회색, 검은색으로 나눌 때, 이는 기존 연구에서 검은 피모색 멧돼지와 하얀 피모색을 가진 랜드레이스 사이에서 나온 F2세대의 표현형의 결과와 유사하였다(Hirooka et al., 2002). 색상 표현형을 두개로 단순화하는 것이, 하나의 표현형을 다른 여러 표현형과 비교하는 것보다 쉬운 것으로 알려져 있다(Visscher et al., 1996). 만약 백색인 표현형을 예로 들어, 백색인 표현형과 비 백색인 표현형들을 비교하는 것이 백색인 표현형과 검은색 표현형을 비교하는 것보다, 어느 색이 영향을 받는 표현형인지 결정하기 어렵기 때문이다.
2. Degree of Colors
Fig 2에서 확인할 수 있듯이 B라인의 밝은 부분인 (143, 148, 122)과, L라인의 밝은 부분인 (132, 136, 117) 간의 RGB 색상 값의 차이가 (11, 10, 5)이다. 그리고 B라인의 어두운 부분인 (36, 37, 31)과 L라인의 어두운 부분인 (24, 25, 21)이 (12, 12, 10)의 차이를 보였다. 두 라인간의 분포는 비슷하지만, 평균 RGB 값은 차이가 났기 때문에 Table 1의 t-test에서 유의한 차이를 보인 것으로 확인되었다.
기존 연구에서 밝은 깃털색 암컷과 어두운 깃털색 수컷의 교배, 밝은 깃털색 수컷과 어두운 깃털색 암컷의 교배에서 유전되는 색은 모두 정규화를 나타내었지만, 밝은 깃털 색의 암컷은 어두운 깃털색 수컷의 교배가 그 반대일 때보다 자손 세대에서 밝은 표현형이 더 많이 유전되었다고 하였다(Dobson et al., 2019). 이는 F1 세대의 결과이고 야생 개체이기에 지역 간의 차이도 존재할 것으로 판단되나, 어미가 밝은 깃털색인 WL인 본 논문의 결과와 유사하였다. 이 결과를 통해 부계의 표현형보다 모계의 표현형이 자손세대에 더 많은 영향을 미쳤다는 사실을 알 수 있다. 또한, 표현형의 색의 정도를 수치화하여 원인 유전자를 찾을 수 있는 가능성을 제시하고 있다.
3. Further Researches
K-means clustering(MacQueen, 1967)이 발표된 이래로 머신 러닝 및 데이터 마이닝 분야에서 다각도로 활용되고 있다. 그 중 k-means clustering에 알려진 오랜 문제 중, 임의로 정해야 하는 k값이 있다. 본 논문에서는 F0의 교배에 사용된 개체의 특징에 따라 2로 나누었지만, F2 개체에서 중간 색상의 개체가 나왔을 때 분류가 쉽지 않은 결과도 얻을 수 있었다. 본 연구에서 YO와 WL을 교배했을 때, 밝은 개체와 어두운 개체만 나오는 것이 아니라 육안으로 확인하기에 갈색인 개체도 있었고, 노란색 깃털색을 가진 개체도 존재했다. 하지만 본 연구에서는 YO와 WL의 특징만을 추출하고 분류하기 위해서 k = 2로 설정하여, 앞에서 기술한 이상 깃털색의 개체를 고려하지 않아 앞으로 추가 연구가 필요할 것으로 판단된다.
K-means clustering의 결과 분포가 7:3이고 대표 RGB 색상이 계산되었다고, 그 그룹을 전부 설명할 수 있는 것은 아니라고 생각한다. 개체에 모든 픽셀의 RGB 평균값에 K-means clustering을 사용했다는 점에서 지나친 일반화의 가능성이 존재한다. 극단적으로 예를 들어, 실험군에 비교될 B와 L라인이 4마리라고 가정하였다. 그 때, 개체 중 RGB 픽셀값이 (100, 100, 100)인 개체와 (0, 0, 0)인 개체가 섞인 분포와, (100, 100, 100) 값의 픽셀과 (0, 0, 0) 값의 픽셀이 50:50으로 혼합되어 있는 개체의 RGB 값의 분포가 같을 수 있기 때문이다.
닭에서 흔히 발견되는 깃털 패턴 중 하나로 횡반이 있다. 횡반 유전인자는 횡반이 아닌 것에 대해 우성이라 반성유전을 한다고 알려져 있다(Oh, 1972). 또한, 본 실험에서 규정한 점박이(spotted) 패턴은 어느 한 영역에 비슷한 값의 RGB가 분포되어 있을 때, 그 주변과 다른 RGB 값의 이상치들은 하나의 속성값으로 계산이 된다(Wandell, 1993). 이런 이유로 표준편차로는 점박이와 횡반 개체를 구분할 수 없다. 추후 연구에서는 점박이와 횡반을 육안으로 분류하고 실험을 진행할 필요가 있다.
사사
본 논문은 농촌진흥청 연구사업(오계와 백색레그혼 교배집단 표현형 정보 활용 육질관련 소재 발굴, PJ0157852022) 및 정보통신기획평가원의 지원을 받은 연구사업(인공지능융합연구센터지원(충남대학교), 2020-0-01441)에 의해 수행되었습니다. 공시된 한국 재래닭의 자료는 국립축산과학원 가축유전자원센터와 연산오계재단에서 제공되었습니다.
REFERENCES
Banerjee S, Zeng L, Schunkert H, Söding J 2018 Bayesian multiple logistic regression for case-control GWAS. PLoS Genetics 14(12):e1007856.

PMid:

PMCid:


Bouma S, Pawley MD, Hupman K, Gilman A 2018 Individual common dolphin identification via metric embedding learning. In: 2018 International Conference on Image and Vision Computing New Zealand (IVCNZ). IEEE, 1-6.


Braquelaire JP, Brun L 1997 Comparison and optimization of methods of color image quantization. IEEE Trans Image Process 6(7):1048-1052.

PMid:


Celebi ME 2011 Improving the performance of k-means for color quantization. Image and Vision Computing 29(4):260-271.


Deng Y, Manjunath B, Kenney C, Moore MS, Shin H 2001 An efficient color representation for image retrieval. IEEE Trans Image Process 10(1):140-147.

PMid:


Dobson AE, Schmidt DJ, Hughes JM 2019 Heritability of plumage colour morph variation in a wild population of promiscuous, long-lived Australian magpies. Heredity 123(3):349-358.

PMid:

PMCid:


Fumihito A, Miyake T, Takada M, Shingu R, Endo T, Gojobori T, Kondo N, Ohno S 1996 Monophyletic origin and unique dispersal patterns of domestic fowls. PNAS 93(13):6792-6795.

PMid:

PMCid:


Hirooka H, De Koning D, Van Arendonk J, Harlizius B, De Groot P, Bovenhuis H 2002 Genome scan reveals new coat color loci in exotic pig cross. J Hered 93(1):1-8.

PMid:


Kasuga H, Yamamoto H, Okamoto M 2000 Color quantization using the fast K‐means algorithm. Syst Comput Jpn 31(8):33-40.


Kerje S, Carlborg Ö, Jacobsson L, Schütz K, Hartmann C, Jensen P, Andersson L 2003 The twofold difference in adult size between the red junglefowl and White Leghorn chickens is largely explained by a limited number of QTLs. Anim Genet 34(4):264-274.

PMid:


Kerje S, Sharma P, Gunnarsson U, Kim H, Bagchi S, Fredriksson R, Schütz K, Jensen P, Von Heijne G, Okimoto R 2004 The Dominant white, Dun and Smoky color variants in chicken are associated with insertion/deletion polymorphisms in the PMEL17 gene. Genetics 168(3):1507-1518.

PMid:

PMCid:


Korte A, Farlow A 2013 The advantages and limitations of trait analysis with GWAS: a review. Plant Methods 9(1):1-9.

PMid:

PMCid:


LeCun Y, Bengio Y, Hinton G 2015 Deep learning. Nature 521(7553):436-444.

PMid:


Lee JH 2019 Researches of the origin and genetic information of Yeonsan Ogye. Korean Poultry Journal 51(3):170-172.

Li Z, Guo R, Li M, Chen Y, Li G 2020 A review of computer vision technologies for plant phenotyping. Comput Electron Agr 176:105672.


Luo W, Xu J, Li Z, Xu H, Lin S, Wang J, Ouyang H, Nie Q, Zhang X 2018 Genome-wide association study and transcriptome analysis provide new insights into the white/red earlobe color formation in chicken. Cell Physiol Biochem 46(5):1768-1778.

PMid:


Mac Queen J 1967 Some methods for classification and analysis of multivariate observations. In: Proceedings of the Fifth Berkeley Symposium on Mathematical Statistics and Probability. Oakland, CA, USA. pp 281-297.

Marini A, Facon J, Koerich AL 2013 Bird species classification based on color features. In: 2013 IEEE International Conference on Systems, Man, and Cybernetics. IEEE. pp 4336-4341.


Mastrangelo S, Cendron F, Sottile G, Niero G, Portolano B, Biscarini F, Cassandro M 2020 Genome-wide analyses identifies known and new markers responsible of chicken plumage color. Animals 10(3):493.

PMid:

PMCid:


Moskvyak O, Maire F, Dayoub F, Armstrong AO, Baktashmotlagh M 2019 Robust re-identification of manta rays from natural markings by learning pose invariant embeddings. In: 2021 Digital Image Computing: Techniques and Applications (DICTA). IEEE, pp 1-8.


Nie C, Zhang Z, Zheng J, Sun H, Ning Z, Xu G, Yang N, Qu L 2016 Genome-wide association study revealed genomic regions related to white/red earlobe color trait in the Rhode Island Red chickens. BMC Genet 17(1):1-7.

PMid:

PMCid:


Oh B-G 1972 Poultry breeding-inheritance of qualitative traits. Korean Poultry Journal 4(6):29-31.

Özdemir D, Akarun L 2002 A fuzzy algorithm for color quantization of images. Pattern Recognition 35(8):1785-1791.


Ozturk C, Hancer E, Karaboga D 2014 Color image quantization: a short review and an application with artificial bee colony algorithm. Informatica 25(3):485-503.


Pérez-Delgado ML 2015 Colour quantization with Ant-tree. Appl Soft Comput 36:656-669.


Scheunders P 1997 A comparison of clustering algorithms applied to color image quantization. Pattern Recogn Lett 18(11-13):1379-1384.


Sertel O, Kong J, Catalyurek UV, Lozanski G, Saltz JH, Gurcan M 2009 Histopathological image analysis using model-based intermediate representations and color texture: Follicular Lymphoma Grading. J Signal Process Syst 55(1):169-183.


Visscher P, Haley C, Knott S 1996 Mapping QTLs for binary traits in backcross and F
2 populations. Genet Res 68(1):55-63.


Wandell BA 1993 Color appearance: The effects of illumination and spatial pattern. PNAS 90(21):9778-9784.

PMid:

PMCid:


Yang CK, Tsai WH 1998 Color image compression using quantization, thresholding, and edge detection techniques all based on the moment-preserving principle. Pattern Recogn Lett 19(2):205-215.