서 론
토종닭은 국내 기후, 풍토에 오랜 기간 적응되어 유지된 고유 유전자원으로, 외래 품종과 구별되는 생리적·형질적 특성을 보유하고 있다(Kim et al., 2019). 토종닭은 국내 축산업에서 향후 품종 개량을 위한 중요 유전자원으로 활용 가능하다는 점에서 보존의 필요성이 꾸준히 강조되어 왔다(Kim et al., 2022; Choi et al., 2025). 특히, 이러한 토종 유전자원은 역사적·환경적 요인을 반영한 독특한 유전자 구성을 유지하고 있어, 충분한 유전적 다양성 확보가 필요하다. 따라서 재래종 토종닭 집단의 유전적 다양성을 체계적으로 평가하고 근친 증가 여부를 지속적으로 감시하는 것은 유전자원 보존 및 육종 체계를 구축하는 데 필수적이다.
유전적 다양성은 가축 집단이 질병이나 환경 변화에 적응하고 생산성과 건강을 유지하는 데 필수적인 기반이다(DeWoody et al., 2021). 그러나 특정 형질에 대한 집약적 선발과 폐쇄된 계통 내 번식은 한정된 유전자풀만 활용함으로써 유전 변이 감소와 근친도 증가를 초래한다(Weigel, 2001). 실제로 닭의 상업 계통에서는 조상 품종에 존재하던 유전 변이의 절반 이상이 유실된 것으로 보고되었다(Muir et al., 2008). 한편, 집단 규모가 작은 품종의 경우 유전적 부동에 따른 다양성 손실과 근친교배 위험이 높기 때문에 생존과 적응력 확보를 위해 유전적 다양성 보존이 특히 중요하다(Palinkas-Bodzsar et al., 2020). 따라서, 분자유전학적 분석법을 이용해 개체군의 다양성 수준과 근친도를 주기적으로 평가하는 체계를 도입하면 혈통 정보가 부족한 경우에도 유전적 다양성 지표를 객관적으로 모니터링할 수 있어 유전자원 관리에 효과적인 것으로 평가되고 있다(Mészáros et al., 2015).
최근 SNP chip 기반 유전체 분석 기술은 집단의 유전적 구조와 다양성을 정밀하게 측정할 수 있는 도구로 널리 활용되고 있다. 본 연구에서는 SNP chip 데이터를 이용하여 재래종 토종닭 4계통의 유전적 다양성 수준을 평가하고, 현재 운영 중인 선발·교배 체계가 근친 증가를 효과적으로 억제하고 있는지 확인하고자 하였다. 또한, 계통 간의 유전적 관계를 분석함으로써, 각 계통의 분화 수준을 유전체 관점에서 규명하고자 하였다.
재료 및 방법
본 연구는 국립축산과학원 가금연구센터에서 보존 중인 재래종 토종닭 4계통을 대상으로 수행하였다. 공시 동물은 회갈색 계통(Grey brown; G), 백색 계통(White; W), 흑색 계통(Black; L), 오계(Ogye; O) 계통으로 4계통이 활용되었다. 공시 동물의 현행 보존 체계는 다음과 같다. 각 계통은 600수 내외로 유지하고 있으며, 매 세대 암컷 약 210수와 수컷 약 50수를 무작위 선발하여 교배하고, 수컷 정액을 혼합해 인공수정하는 방식으로 이루어지고 있다. 이러한 관리 체계는 특정 개체에 의한 유전적 편향성을 억제함으로써 집단 내 다양성을 유지하는 데 목적이 있지만, 실제로 유전체 수준에서 다양성이 안정적으로 유지되고 있는지에 대한 평가는 제한적으로 이루어져 왔다. 폐쇄 집단에서는 세대가 반복될수록 근친이 누적될 가능성이 존재하므로, 집단유전학적 지표를 활용한 정량적 검토는 현행 교배 체계의 적절성을 평가하는 데 중요한 근거가 된다(Wright, 1965; Hartl and Clark, 1989).
본 연구를 위해 각 계통 당 48수씩 총 192수를 실험에 이용하였다. 각 개체에 대해 날개정맥에서 혈액을 채혈하여 EDTA 처리된 튜브에 보관하였고, 채혈 후 단기간 냉장 보관한 뒤 DNA 추출 시에 활용되었다. 동물실험은 국립축산과학원 동물실험윤리위원회(IACUC)의 승인된 동물실험방법(승인번호: NIAS2022-0568)에 따라 동물복지 지침을 준수하여 수행하였다.
Genomic DNA는 상업용 혈액 DNA 추출 키트(MagextractorTM, TNT Research, Korea)를 이용하여 제조사 매뉴얼에 따라 추출하였다. 추출된 DNA의 농도와 순도는 분광광도계(BioTek Epoch Microplate Spectrophotometer, Agilent Technologies, USA)를 사용하여 확인하였다. 유전자형 분석은 Illumina 플랫폼 기반의 custom 가금 30K SNP chip을 이용하여 수행하였다.
유전 변이와 분석의 신뢰도를 확보하기 위하여 SNP chip 자료에 대해 품질관리를 수행하였다. 개체 수준에서는 전체 SNP에 대한 genotyping rate가 0.90 미만인 개체를 제외하였으며, 본 연구에서 모든 개체가 기준을 만족하여 추가적인 개체 제거는 이루어지지 않았다. 마커 수준에서는 전체 계통 데이터셋에서 minor allele frequency가 0.01 미만인 SNP와 genotyping rate가 0.90 미만인 SNP를 제거하였다. 그 결과, 초기 30,816개 SNP 중 저빈도 변이 및 결측률이 높은 마커를 제외한 28,599개의 SNP를 최종 분석(계통별 각 48수)에 이용하였다. 모든 전처리 과정에는 PLINK v1.9 소프트웨어를 이용하였다(Purcell et al., 2007).
계통별 유전적 다양성 평가를 위해 관측 이형접합도(Hobs)와 기대 이형접합도(Hexp)를 계산하였다. 계산을 위해 R software의 adegenet 패키지를 이용하였으며(R Core Team, 2021), 산출된 Hobs와 Hexp를 바탕으로 Wright(1965)의 정의에 따라 근친계수 FIS를 아래의 식을 적용해 계산하였다.
계통별 FIS는 각 마커에 대한 FIS 값을 평균하여 구하였다. 4계통(G, W, L, O)의 Hobs, Hexp 및 FIS 값을 비교함으로써 계통 내 유전적 다양성 수준과 근친의 정도를 평가하였다.
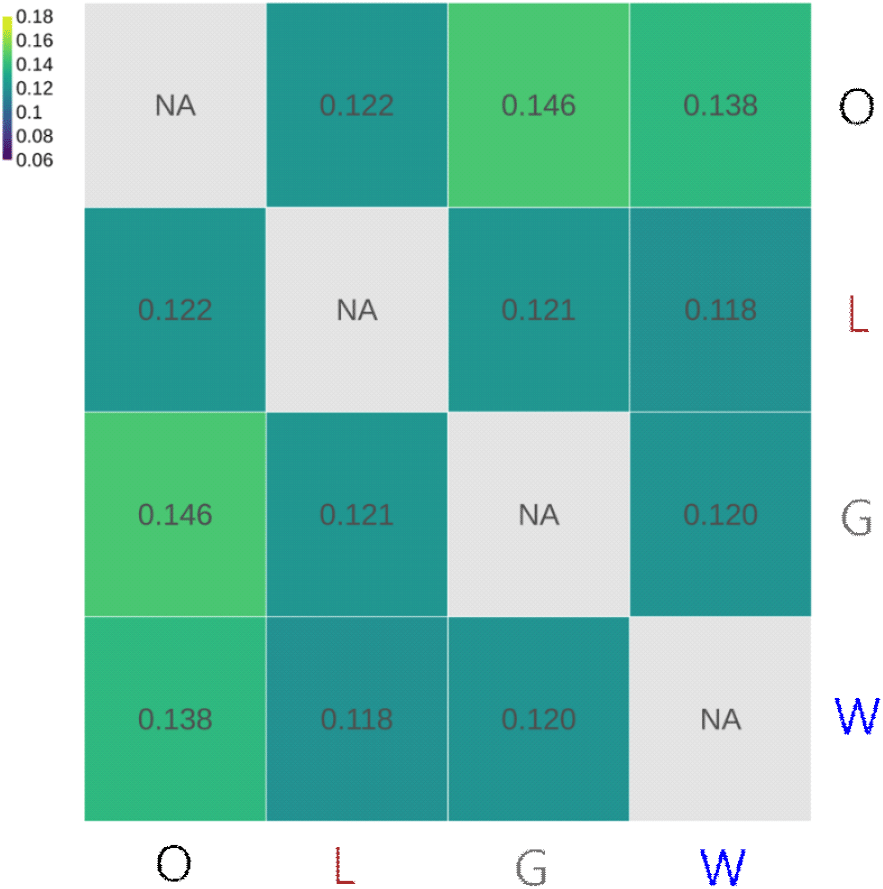
계통 간 유전적 분화 정도는 FST 값으로 정량화하였다. 이를 위해 R의 hierfstat 패키지를 사용하여 전체 데이터셋에서 Weir & Cockerham 방식의 계통 간 pairwise FST 값을 산출하였으며, R에서 heatmap과 함께 시각화하였다.
계통별 데이터셋을 이용해 PLINK v1.9를 활용하여 SNP 쌍 간 상관계수(r2) 값을 계산하였다. 계산 시, 윈도우 크기를 5 Mb로 설정하여 윈도우 내의 모든 SNP 쌍의 r2를 산출하였으며, r2는 다음의 식을 활용하여 계산되었다(Lee et al., 2017).
Ne는 LD 기반 추정 방법을 이용하여 계통별로 산출하였으며, 추가적으로 향후 50세대 동안 변화하는 이형접합도의 변화 추이 예측값을 도출하였다. Ne는 아래의 식을 활용하여 계산되었다.
특정 세대(t)에서의 Ne는 아래의 식을 활용하여 계산되었다.
C는 마커 간 유전적 거리를 의미하며 Morgan 단위로 표현된다. 이 공식은 마커 간 물리적 거리에 따라 관찰되는 LD가 서로 다른 시기의 재조합을 반영한다는 가정에 기반한다. 즉, 서로 가까운 위치에 있는 마커 사이의 r2는 최근 세대에서 발생한 재조합 신호를 주로 반영하는 반면, 보다 멀리 떨어진 마커 간 r2는 더 과거의 역사적 재조합 패턴을 반영하는 것으로 간주된다(Lee et al., 2017). 분석은 R software에서 수행되었다.
집단구조 분석을 위해 전체 계통 데이터셋을 대상으로 PLINK v1.9를 이용하여 주성분분석을 수행하였고, R을 이용해 시각화하였다.
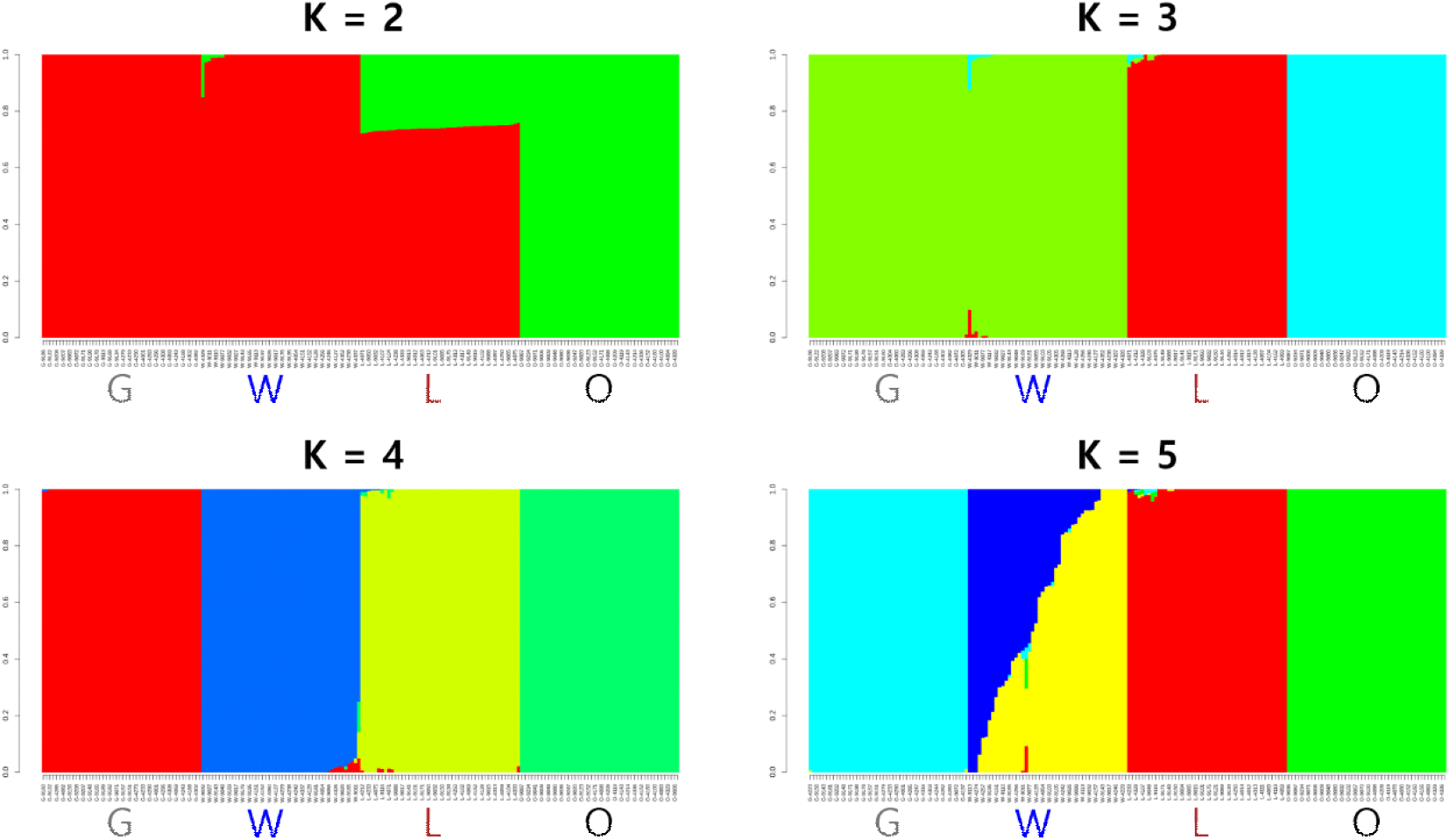
계통 간 조상집단 혼합 정도를 확인하기 위해 ADMIXTURE v1.3.0을 이용하여 전체 계통 데이터셋에 대한 admixture 분석을 수행하였다(Alexander et al., 2009). 군집 수 K는 2에서 10까지 변화시키며 교차검증(CV) 오류 값을 비교하였고, CV 오류가 최소가 되는 값을 최적 군집 수로 선택하였다. 선택된 K 값에 대해 각 개체의 admixture 비율을 계산하고, 이를 R에서 막대그래프 형태로 시각화하여 계통 간 유전적 혼합 및 분리 양상을 해석하였다.
유전적 거리 및 계통수 분석은 전체 계통 및 각 계통별 데이터셋으로 수행하였다. 먼저 PLINK v1.9를 이용하여 유전자형 자료 기반 개체 간 유전적 거리를 산출하였다. 산출된 거리행렬을 이용해 R의 ape 패키지를 사용하여 neighbor-joining 알고리즘으로 계통수를 작성하였으며, pheatmap 패키지를 이용하여 동일한 거리행렬 기반 heatmap을 작성하였다.
결과 및 고찰
한국 토종닭 4계통의 유전적 다양성 지표로 관측 Hobs와 Hexp를 계통별로 비교한 결과, 4계통 모두 Hobs와 Hexp 값이 거의 유사하게 나타났다. 구체적으로 G 계통의 Hexp = 0.241, Hobs = 0.246(FIS = −0.021), W 계통 Hexp = 0.250, Hobs = 0.257(FIS = −0.028), L 계통 Hexp = 0.284, Hobs = 0.296(FIS = −0.042), O 계통 Hexp = 0.251, Hobs = 0.254(FIS = −0.012)로 측정되었다(Fig. 1). 관측치가 기대치보다 높아 FIS 값이 −0.01에서 −0.04로 거의 0에 가까운 작은 음수를 보였는데, 이는 계통 내 근친교배로 인한 이형접합도 감소 현상이 거의 없음을 의미한다(Wright, 1965). 일반적으로 FIS 값이 0에 가까울수록 집단 내 근친 정도가 낮음을 나타내며, FIS>0.25일 경우 매우 높음 수준의 근친도가, 0.15~0.25는 높음, 0.05~0.15는 보통, 0~0.05는 낮음으로 분류된다(Hartl and Clark, 1989). 본 계통들의 FIS는 음수를 띠었으며, 이는 최근에 적용된 교배 방식에 의해 집단의 이형접합도가 증가하는 양상이 존재했음을 반영한다. 이를 통해, 근친 회피를 통한 유전적 다양성이 유지되고 있음을 알 수 있다(Strillacci et al., 2017). 한편 4계통의 Hexp(0.24~0.28 수준)는 다른 토종 닭 집단들의 보고치에 비해 낮은 편으로, 이는 오랜 기간 폐쇄된 계통으로 선발·육종되어 온 결과 전체 대립유전자 다양성은 다소 제한되었음을 나타낸다(Zhang et al., 2020). 그럼에도 불구하고 Hobs가 Hexp와 크게 다르지 않다는 점에서, 현재 각 계통 내 유전적 다양성이 안정적으로 유지되고 있음을 확인할 수 있었다.
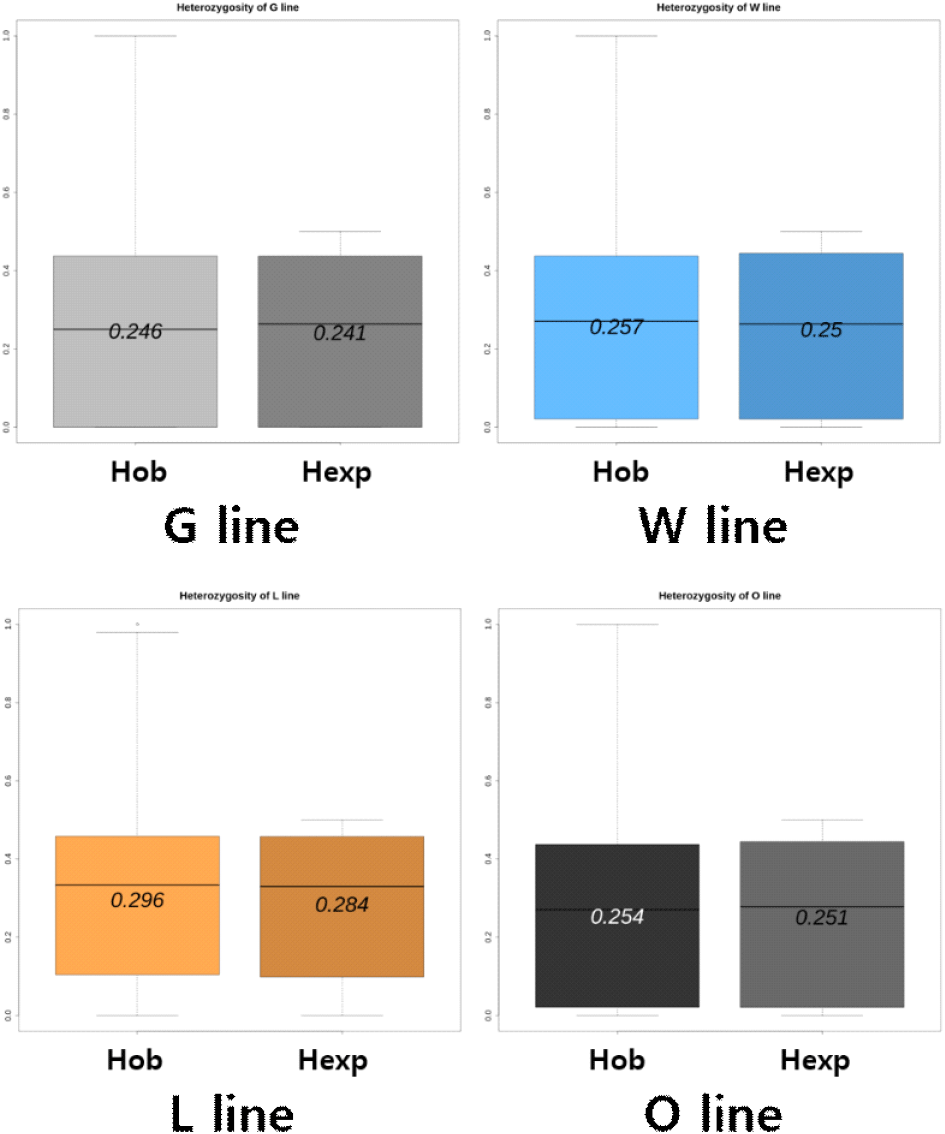
Fig. 2에 나타난 4계통의 LD 분석 결과, 모든 계통에서 유사한 LD 감소 양상이 관찰되었다. 각 계통 내 SNP 간 r2를 물리적 거리별로 산출한 결과, 인접한 위치에서는 평균 r2 값이 약 0.4 수준으로 비교적 높았으나 거리가 증가함에 따라 감소하여 약 100 kb 이내 거리에서 r2가 0.2 미만으로 떨어졌고, 1.5 Mb 이상 떨어진 마커들 간에는 r2가 0.1 내외의 낮은 수준까지 감소하였다. 이러한 LD decay 경향은 4계통 모두에서 비슷하게 나타났으며, 계통 간 큰 차이는 관찰되지 않았다. 이러한 경향은 이전의 한국 토종닭 계통 연구 결과와도 유사한데, Seo et al.(2018)이 보고한 황갈색, 적갈색 계통의 LD 수준이 본 연구 계통들의 LD와 유사하며, 이는 한국 토종닭 재래종 계통들이 전반적으로 낮은 LD 수준을 보임을 뒷받침한다.
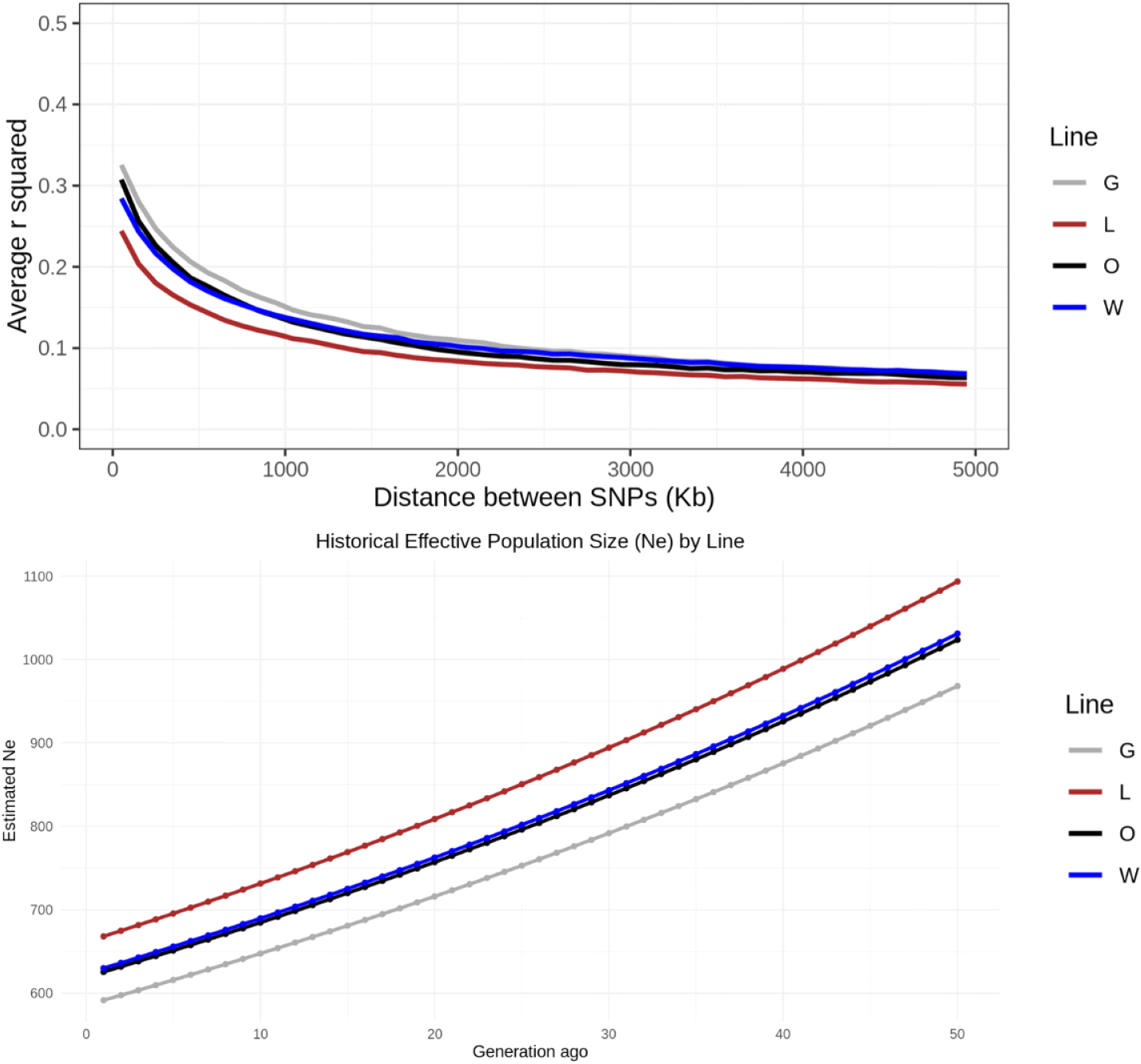
LD 분석을 바탕으로 추정한 Ne는 계통별로 크게 다르지 않은 값을 나타냈다. 현재 세대(t = 0 기준)의 Ne는 L 계통에서 약 668로 가장 높았으며, W 계통 630, O 계통 625, G 계통 591 순으로 추정되었다. 일반적으로 Ne = 50 미만이면 단기 절멸 위험이 있는 취약 집단, Ne = 500 이상이면 장기적으로 유전적 다양성을 유지할 수 있는 집단으로 간주된다(Franklin, 1980). 본 연구에서는 모든 계통의 Ne가 약 600 내외의 값을 보여주었으며, 이는 집단 내 유전적 다양성이 안정적으로 유지되고 있음을 의미한다.
계통별 계통수 분석을 통해 각 계통 내부의 유전적 구조를 확인한 결과, 개체들 사이에 뚜렷하게 분리되는 군집 형성은 관찰되지 않았다(Fig. 3). 이는 각 계통 내 개체들이 계통 단위로 동질적인 집단을 형성하고 있음을 보여준다. 이는 앞서 언급한 Hobs/Hexp 분석에서 확인된 바와 같이 계통 내 이형접합도가 고르게 유지되고 있는 결과와 부합한다. 또한, 특정 형매 관계가 집단에 지배적으로 관찰되지 않았으며, 이를 통해 현행 교배 체계가 적정 수준의 무작위성을 확보하고 있다고 판단된다.
계통 간 유전적 관계를 확인하고자 수행된 전체 계통 데이터셋을 이용한 주성분분석 결과, 계통에 따른 명확한 군집화가 확인되었다(Fig. 4). 특히, PC1~PC3 상에서 시각화한 결과 모든 개체들이 계통별로 구분되는 클러스터를 형성하였다.
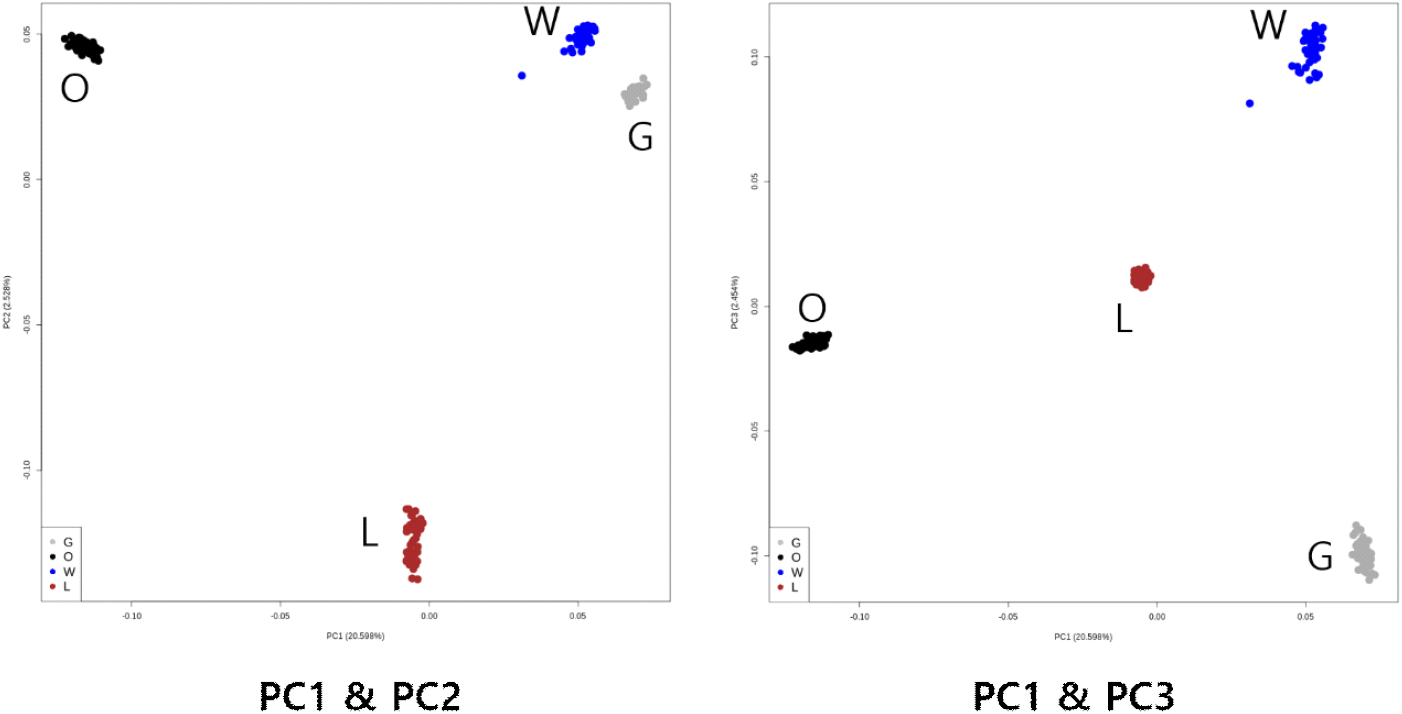
Fig. 5에 나타난 ADMIXTURE 분석을 통해 계통 간 유전적 구성비를 추정한 결과, 가장 적합한 군집수 K = 4로 도출되었다. K = 4에서 계통 간 유전적 혼입이 거의 발견되지 않았다. K = 2일 때 O 계통이 단독 군집을 형성하여 다른 3계통(G, W, L)과 완전히 분리되었으며, K = 3에서는 추가로 L 계통이 분리되어 W와 G 계통이 동일 군집으로 묶이는 양상이 나타났다. 이러한 계통 간 분리 순서는 앞서의 계통수 결과와 주성분분석 결과에서 관찰된 패턴과 일치한다. 즉, O 계통이 가장 이질적으로 먼저 분화되고, 다음으로 L 계통이 G-W 계통군과 분리되며, G 계통과 W 계통은 가장 유사하게 남는 구조가 확인되었다. 실제 집단 구조분석을 수행한 다른 가축 대상 연구에서도 유사하게 유연관계가 가까운 품종들끼리는 낮은 K 값에서 동일 군집으로 묶이다가 더 높은 K에서 분리되는 양상이 보고된 바 있다(Sinhalage, 2023).
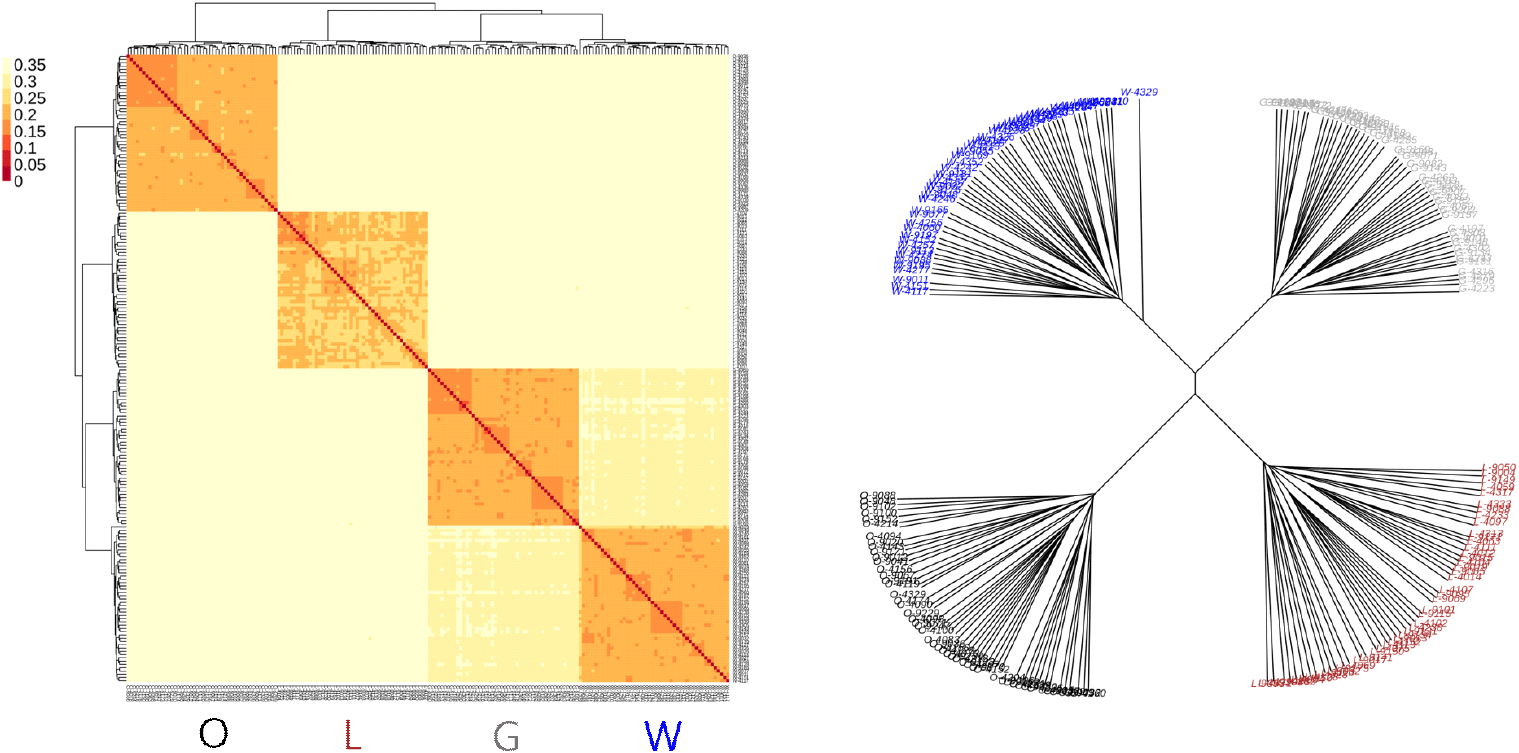
Fig. 6에 나타난 전체 계통의 유전적 거리 기반 계통수 및 heatmap 분석 결과, PCA 및 Admixture에서 나타난 계통 간 관계가 일관되게 관찰되었다. 개체들은 계통별로 명확히 분기되어 군집을 형성하여 계통 간 유전적 차이를 보여주었다. W 계통과 G 계통이 가장 근접한 관계를 보였으며, 이후 L 계통이 합쳐지며, O 계통이 가장 멀리 분리되는 형태를 보였다. 계통 간 평균 유전적 거리 값을 정량적으로 보면, 가장 가까운 G-W 계통 간 거리는 가장 가까운 반면, O 계통과 G 또는 W 계통 간 거리가 가장 멀었다(Table 1).
| Line 1 | G | O | W | L |
|---|---|---|---|---|
| G | - | 0.3555 | 0.317 | 0.3382 |
| O | 0.3555 | - | 0.3504 | 0.3482 |
| W | 0.317 | 0.3504 | - | 0.3396 |
| L | 0.3382 | 0.3482 | 0.3396 | - |
마지막으로, 계통 간 유전적 분화 정도를 Wright의 FST 값으로 평가하였다. Fig. 7에 나타낸 것처럼, 4계통의 pairwise FST 값을 산출한 결과, 0.11<FST<0.15 범위에 분포하여 중간 정도의 분화 수준을 보였다. 가장 FST 값이 낮은 계통 쌍은 W 계통과 L 계통으로 0.118이었고, 가장 높은 쌍은 O 계통과 G 계통 및 O 계통과 W 계통 조합으로 약 0.13~0.15 수준이었다. Wright의 기준에 따르면 FST 값이 0.05 미만은 낮은 수준, 0.05에서 0.15 사이는 중간 수준, 0.15에서 0.25 사이는 높은 수준, 0.25 초과는 매우 높은 수준의 집단 분화를 의미한다(Wright, 1965; Hall, 2022).
본 연구는 무작위 선발과 정액 혼합을 기반으로 한 현행 교배 체계가 폐쇄 집단 내 근친도 증가를 효과적으로 억제하고 유전적 다양성 유지에 기여하고 있음을 유전체 수준에서 확인하였다. 특히, 토종닭 유전자원을 보유한 소규모 농가 및 민간 육종 회사의 경우, 본 연구에서 검토한 선발·교배 방법을 적용한다면 유전적 다양성 유지 등 효율적인 집단 관리 전략이 될 수 있을 것으로 사료된다. 따라서 본 결과는 토종닭과 같은 국내 가금 유전자원의 지속적 보존 및 활용을 위해, 현행 교배 체계가 유효한 기본 관리 모델이 될 수 있음을 시사한다.
적 요
본 연구는 한국 토종닭 재래종 4개 계통을 대상으로 30K SNP chip 자료를 이용하여 계통 내 유전적 다양성과 계통 간 유전적 구조를 평가하였다. 기대이형접합도와 관측이형접합도는 4계통에서 유사한 수준을 보였고, 근친계수(FIS)는 모두 음의 값을 나타내 근친 증가가 발생하지 않았음을 확인하였다. 연관불평형 감소 양상은 계통 간 큰 차이가 없었으며, 연관불평형 기반 유효집단 크기는 최근 세대에서 590~670 범위를 보여 안정적인 다양성 유지가 이루어지고 있었다. 주성분분석, 집단 구조 분석 및 계통수 분석에서는 4계통이 명확히 분리되었으며, 오계 계통이 가장 독립적인 구조를 보였고 회갈색 계통과 백색 계통이 상대적으로 가까운 유전적 관계를 나타냈다. 이러한 결과는 현행 선발 및 교배 체계가 각 계통의 다양성을 적절히 유지하면서 계통 간 유전적 차이를 안정적으로 유지하고 있음을 시사한다.