서 론
최근 지구온난화로 인한 이상기후 현상이 빈번해지면서, 고온 환경은 가축의 생산성과 생존에 심각한 영향을 미치고 있다. 특히 땀샘이 없어 체온 조절이 어려운 가금류는 고온 스트레스에 취약하여 산란율 감소, 면역력 저하, 폐사율 증가 등 다양한 문제를 초래한다(Guo et al., 2022). 이러한 피해는 가축의 생리적 건강과 복지 수준을 저하시킬 뿐만 아니라, 축산업의 생산성 및 경제적 손실로 이어질 수 있다. 따라서 열내성 반응의 유전적 기초를 규명하는 것은 기후 변화에 대응한 지속 가능한 축산을 위한 핵심 과제이며, 고온 적응형 품종의 개발은 산업적·생태적 측면에서 모두 중요하다.
한국 재래닭(Korean native chicken, KNC)은 약 2,000년 전부터 한반도에 존재해 온 재래종과, 국내에서 7세대 이상 사육되며 토착화된 외래종을 포괄하며, 복원·선발 과정을 거쳐 유지되고 있는 유전자원이다(Jin et al., 2017; Lee et al., 2018). KNC는 외래 품종과의 교잡 없이 고유한 형질을 유지해왔음에도 불구하고 이들의 국내 생산량은 2024년도 기준 전체 육계 생산량의 2.5%로 매우 낮으며, 국내에 유통되는 실용 산란계 및 육계의 종계는 대부분 외래종으로 구성되어 있다(Animal and Plant Quarantine Agency, 2025). 이러한 재래닭의 낮은 자급률은 국내 축산업의 지속가능성과 식량주권 측면에서 잠재적 위협 요인으로 작용하고 있어, KNC의 유전적 다양성과 고온 적응 형질을 과학적으로 규명하고 이를 산업적 가치로 전환하기 위한 연구가 필요하다.
이에 본 연구는 재래닭의 고온 환경 적응과 관련된 유전적 기반을 규명하고, 이를 통해 KNC의 열내성 향상을 위한 육종 전략 수립에 기초자료를 제공하는 것을 목적으로 수행되었다. 이를 위해 열내성과 연관된 19개 후보 유전자 구간의 단일염기다형성(single nucleotide polymorphism, SNP) 변이를 분석하여 KNC, 열대 지역 재래닭, 그리고 실용계 집단 간의 유전적 다양성과 집단 구조 차이를 비교하였다. 집단 간 유전적 차이를 다각적으로 평가하기 위해 principal component analysis (PCA)를 통해 전체 변이에서 나타나는 군집 패턴을 확인하고, fixation index (FST) 분석으로 집단 간 분화 수준을 정량적으로 산출하였으며, Nei 유전거리를 기반으로 계통수 분석을 수행하여 집단 간 계통적 관계를 구조적으로 검토하였다. 이러한 통합 분석을 통해 KNC의 열내성 관련 유전적 특징을 외래 집단과 비교하여 종합적으로 규명하고자 하였다.
재료 및 방법
본 연구는 KNC 6개 계통(회갈색, 흑색, 적갈색, 황갈색, 백색, 연산오계)에서 계통별 10수씩 총 60수, 열대 지역 재래닭(인도네시아, 방글라데시, 스리랑카, 태국, 베트남) 50수, 실용계 집단(이탈리아, 이란, 미국의 육계 및 산란계) 19수를 포함하여 총 129수의 개체를 대상으로 수행하였다(Table 1).
모든 개체의 유전체 정보는 공개 데이터베이스에 제공된 차세대 염기서열 분석(next-generation sequencing, NGS)자료를 활용하였다. KNC 데이터는 농촌진흥청 농업생명공학정보센터(National Agricultural Biotechnology Information Center, NABIC)에서 확보하였으며(https://nabic.rda.go.kr/), 열대 지역 재래닭 및 실용계 집단에 대한 정보는 Galbase 데이터베이스로부터 확보하였다(http://animal.omics.pro/code/index.php/ChickenVar).
공개 데이터베이스에서 확보한 두 개의 variant call format (VCF) 파일은 SnpEff (Cingolani et al., 2012) software를 사용하여 Ensembl GRCg6a Release 113을 기준으로 annotation을 수행하였다.
본 연구는 선행문헌에서 열내성과의 연관성이 보고된 유전자들을 대상으로 분석을 수행하였다. 분석 대상 유전자는 열스트레스 조건에서 발현 변화나 기능적 역할이 보고된 경우, 고온 환경 개체군에서 선발 신호가 확인된 경우, 또는 열내성 관련 주요 생물학적 경로에 속하는 경우를 기준으로 선정되었다. 이 기준에 해당하는 총 19개의 후보 유전자(SOCS2, HSPH1, ARPP21, RB1CC1, HSF1, PLCB4, CITED2, HSF3, TSHR, HSP90AA1, HSP70, SIRT1, NFKB2, BAG3, HSP60, PRKCA, HSP25, CDC37, LPAR2) 내 위치하는 SNP 변이를 추출하여 분석에 이용하였다. 각 유전자의 상세 정보는 Table 2에 제시하였다. 추출된 SNP 변이는 bcftools(v1.10.2; Danecek et al., 2021) software를 이용하여 정렬 및 정규화 과정을 수행한 뒤 병합하여 본 연구의 유전적 다양성 및 집단 구조 분석에 활용하였다.
| Gene | Full name | Main function | Physical position | Reference |
|---|---|---|---|---|
| SOCS2 | Suppressor of cytokine signaling 2 | Cytokine-signaling suppressor detected as a selection signal in heat-adapted populations | 1:44921695-44928309 | Walugembe et al. (2019) |
| HSPH1 | Heat shock protein family H (Hsp110) member 1 | Co-chaperone that regulates HSP70 activity, showing increased expression after heat stress | 1:176342009-176365543 | Pritchett et al. (2023) |
| ARPP21 | cAMP regulated phosphoprotein 21 | RNA-binding protein involved in cellular heat response through stress-granule formation and post-transcriptional regulation under heat stress | 2:45360038-45491204 | Zhuang et al. (2020) |
| RB1CC1 | RB1 inducible coiled-coil 1 | Upregulated after heat stress and involved in the inhibition of apoptosis and the regulation of programmed cell death | 2:109934195-110008988 | Luo et al. (2014) |
| HSF1 | Heat shock factor 1 | Key transcription factor activated under heat stress to induce HSP expression | 2:130889350-130994715 | Rachman et al. (2024) |
| PLCB4 | Phospholipase C beta 4 | Involved in immune defense, energy metabolism, and oxidative stress response, and identified as a selection signal in heat-adapted populations | 3:14057963-14274875 | Asadollahpour Nanaei et al. (2022) |
| CITED2 | CBP/p300-interacting transactivator with ED-rich tail 2 | Transcriptional co-activator that protects cells from heat stress by inhibiting hypoxia-induced apoptosis | 3:54058268-54075055 | Wang et al. (2015) |
| HSF3 | Heat shock factor 3 | Key heat-responsive transcription factor activated by severe heat stress to induce HSP expression | 4:281393-297917 | Kawazoe et al. (1999) |
| TSHR | Thyroid stimulating hormone receptor | Regulates energy metabolism and thermoregulation through thyroid hormone signaling, and identified as a selection signal in heat-adapted populations | 5:40972483-41022453 | Guo et al. (2022) |
| HSP90AA1 | Heat shock protein 90 kDa alpha, class A member 1 | Regulates protein folding and signaling stability, and protects cells from heat stress | 5:49677834-49686470 | Wang et al. (2013) |
| HSP70 | Heat shock protein 70 | Heat-induced chaperone that maintains protein folding and protects cells by inhibiting apoptosis | 5:53057329-53061067 | Balakrishnan et al. (2023) |
| SIRT1 | Sirtuin 1 | Regulation of oxidative stress and DNA stability contributing to antioxidant-mediated cellular protection under heat stress | 6:7313578-7338123 | Kishawy et al. (2023) |
| NFKB2 | Nuclear factor kappa B subunit 2 | Inflammation and ROS regulation for cellular protection under stress | 6:17766967-17776582 | Yao et al. (2025) |
| BAG3 | BCL2-associated athanogene 3 | Co-chaperone modulating HSF1 interactions and heat-induced nuclear shuttling for stress adaptation | 6:31227774-31248047 | Jin et al. (2015) |
| HSP60 | Heat shock protein 60 | Heat-induced upregulation supporting protein refolding, mitochondrial stability and cellular stress protection | 7:10214905-10231730 | Yan et al. (2009) |
| PRKCA | Protein kinase C alpha | Kinase linked to heat tolerance through prolactin signaling and oxidative stress regulation | 18:7314263-7428828 | Fleming et al. (2016) |
| HSP25 | Heat shock protein 25 | Strongly heat-induced small chaperone mediating aggresome dissociation and cellular protection | 19:4402072-4406049 | Wang et al. (2013) |
| CDC37 | Cell division cycle 37 | HSP90 co-chaperone involved in stabilizing and refolding heat-denatured proteins and facilitating damaged-protein clearance | 30:168682-178823 | Rachman et al. (2024) |
| LPAR2 | Lysophosphatidic acid receptor 2 | Elevated after heat stress and associated with cell survival and spermatogenesis signaling | 30:1127965-1137777 | Wang et al. (2015) |
SNP 변이에 대한 품질관리(quality control, QC)는 PLINK 2.0 (Chang et al., 2015) 소프트웨어를 사용하여 수행하였다. QC 기준은 Hardy-Weinberg equilibrium (HWE) P-value<1 × 10−7 (--hwe 1e-7 midp), 유전자형 결측률 10% 미만 (--geno 0.1), minor allele frequency (MAF)≥0.05 (--maf 0.05)으로 설정하였으며, 이러한 조건을 충족한 SNP만을 이후 분석에 사용하였다.
집단 간 유전적 변이를 요약하여 집단 구조 및 군집 패턴을 확인하기 위해 PCA 분석을 실시하였다. PCA는 대규모 SNP 데이터에서 고차원 변수를 소수의 주요 분산 축(principal components, PCs)으로 축소하여, 유전적 다양성 및 집단 간 분화 패턴을 시각화하는 데 사용되는 통계적 방법이다(Abdi and Williams, 2010). 본 연구에서는 PLINK 2.0 소프트웨어를 활용하여 PCA를 수행하였으며, 산출된 고유벡터(eigenvectors)를 기반으로 R software(R Core Team, 2025)에서 개체 간 유전적 분포 양상을 시각화하였다. 또한, 집단 간 분포 경향을 파악하기 위해 KNC 각 집단별로 95% 신뢰타원(ellipse)을 적용하였다.
집단 간 대립유전자 빈도 차이를 정량화하여 유전적 분화 수준을 평가하기 위해 FST 분석을 수행하였다. FST 분석은 전체 유전변이 중 집단 간에 존재하는 분화의 비율을 나타내는 지표로, 값이 높을수록 집단 간 유전적 차이가 크다는 것을 의미한다(Meirmans and Hedrick, 2011). 본 연구에서는 Weir and Cockerham (1984)의 방법을 적용하여 R software의 “SNPRelate” 패키지(Zheng, 2013)를 사용해 pairwise FST 값을 산출하고 이를 heatmap으로 시각화하였다.
집단 간 유전적 유사성과 차이를 비교하여 계통적 관계를 규명하기 위해 유전적 거리를 기반으로 한 계통수 분석(phylogenetic tree)을 수행하였다. 계통수는 집단 간 유전적 변이 정보를 이용하여 진화적 분화와 유사성의 정도를 시각적으로 표현하는 방법으로, 유전적으로 유사한 집단일수록 더 가까운 가지로 연결된다. 본 연구에서는 Nei 유전거리(Nei’s genetic distance; Nei, 1972)를 산출한 뒤, neighbor-joining 방법(Saitou and Nei, 1987)을 적용하여 phylogenetic tree를 구성하였으며, 모든 분석은 R software 환경에서 수행하였다.
결과 및 고찰
KNC와 외래 집단 간의 열내성 연관 유전적 다형성 및 집단 구조 분석을 위해, 선행연구를 통해 선정된 19개 유전자 영역의 SNP를 추출하고 QC 과정을 거쳐 총 8,197개의 SNP를 분석에 활용하였다.
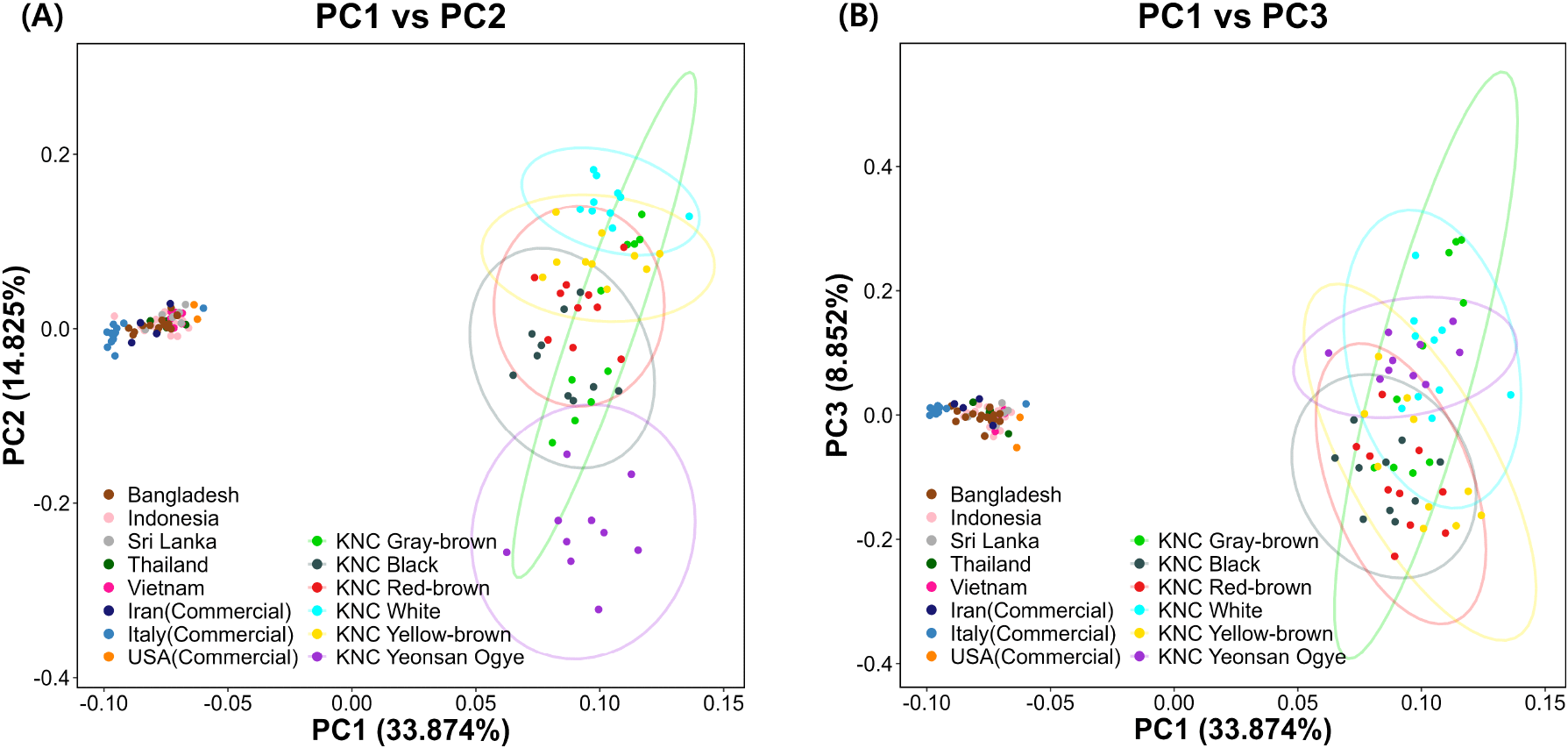
집단 간 유전적 구조 차이를 파악하기 위해 PCA를 수행한 결과, PC1은 33.87%의 변동을 설명하며 가장 큰 비중을 차지하였으며, PC2와 PC3는 각각 14.82%와 8.85%로 확인되었다(Fig. 1). 상위 세 개의 주성분이 전체 변동의 약 57.6%를 설명하여, 이들이 집단 간 유전적 차이를 반영하는 주요 축으로 기능함을 확인하였다. 특히, PC1 축에서 KNC와 외래 집단이 명확하게 분리되었으며, 이는 열내성 연관 유전자 구간에서 두 집단 간 유전적 구조 차이가 있을 가능성을 시사한다. PC2와 PC3 축에서 외래 집단은 하나의 클러스터를 형성하는 것과 달리, KNC는 계통별로 각각의 클러스터를 형성하여 집단 내 높은 유전적 다양성을 보유하고 있음을 시사하였다.
PCA에서 확인된 집단 간 분화 양상을 보다 정량적으로 평가하기 위해 FST 분석을 수행하였다(Fig. 2). 분석 결과, KNC는 열대 지역 재래닭 및 실용계 집단과 비교하여 0.14~ 0.34 범위의 FST 값을 보여, PCA에서 나타난 집단 간의 구분과 일치하는 뚜렷한 유전적 분화를 나타냈다. 이는 KNC가 다른 집단과 구별되는 열내성 연관 유전적 특성을 보유하고 있음을 뒷받침한다.
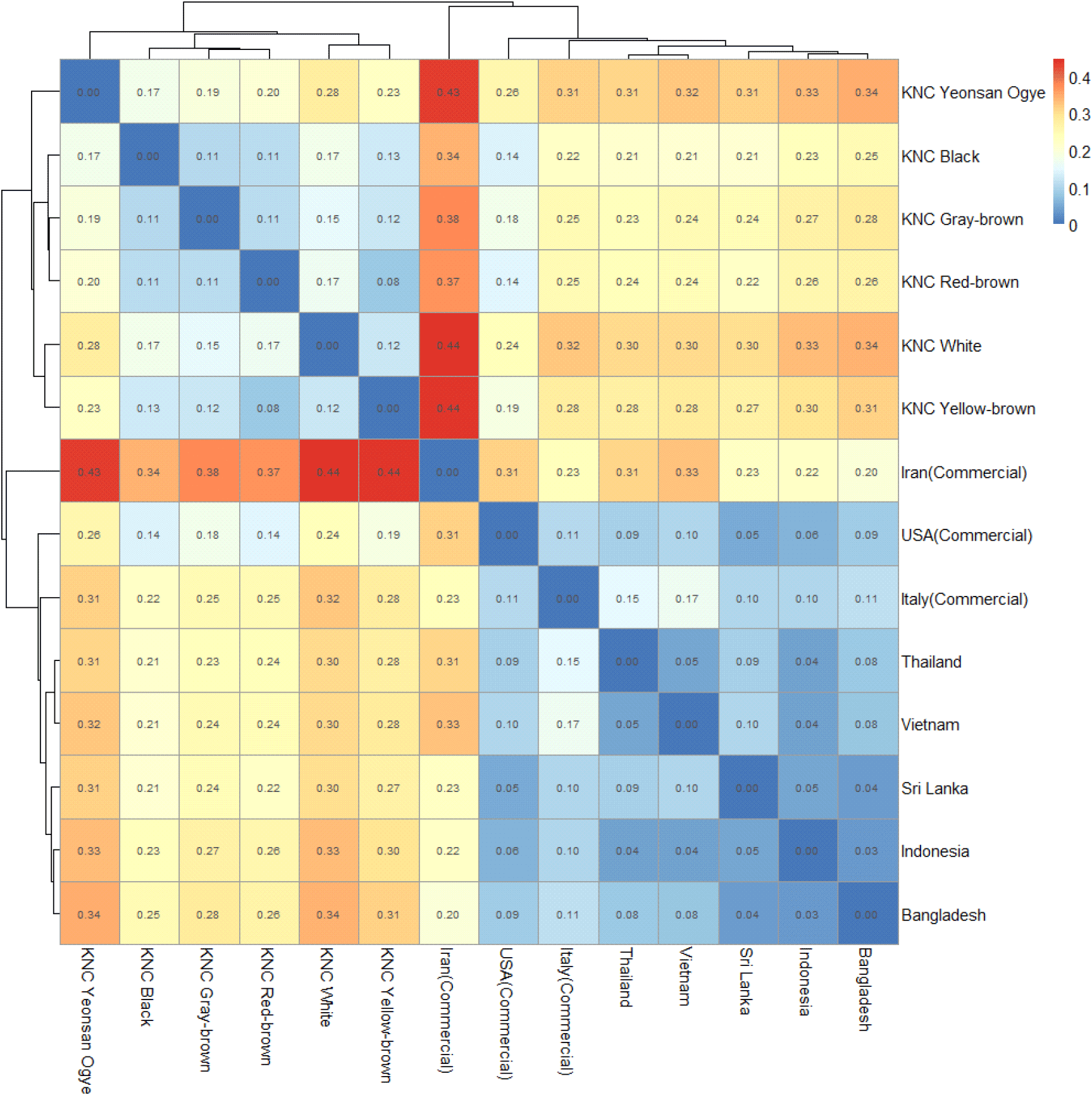
KNC 집단 내에서는 연산오계를 제외한 5개 계통에서 0.08~0.17의 낮은 분화도가 관찰되어, KNC 계통 간 높은 유전적 유사성을 보였다. 반면, 연산오계는 다른 KNC 계통과 0.17~0.28 범위의 FST 값을 보여 KNC 집단 내에서도 독립적인 유전적 구조를 가진 계통임을 나타냈다. KNC 계통 간 낮은 분화도는 기존 연구에서도 일관되게 보고되었으며, 초위성체(microsatellite) 마커 분석에서도 여러 KNC 계통이 하나의 근접한 군집으로 분류되는 양상이 확인되었다(Seo et al., 2015). 이러한 군집 구조는 KNC가 제한된 규모의 개체군을 기반으로 복원되어 장기간 유사한 보존·번식 체계로 유지된 관리 체계에 기인하여, 계통별 유전적 분화가 크게 축적되지 않은 것으로 해석된다.
외래 집단 간 비교에서는 이탈리아 및 미국 실용계 집단은 열대 지역 재래닭과의 FST 값이 0.05~0.17 범위로 낮게 나타나 열대 계통과 유전적으로 유사함이 확인되었다. 반면, 이란 실용계 집단은 열대 재래닭과는 0.20~0.33, KNC와는 0.34~0.44의 FST 값을 보여 상대적으로 높은 수준의 유전적 분화도를 보였다. 이는 이란 계통이 외래·열대 집단과 비교하여도 독립적인 유전적 구조를 보유하고 있으며, 특히 KNC와는 뚜렷하게 구분되는 집단임을 시사한다.
집단 간 유전적 관계를 평가하기 위해 Nei 유전거리를 산출하고, 이를 기반으로 phylogenetic tree를 구성하였다(Fig. 3). 분석 결과, 모든 KNC 집단은 하나의 공통 가지에서 분리되며 집단 간 높은 근연관계를 나타내었으나, 외래 집단과는 서로 명확히 구분되는 가지를 형성하였다. 이는 두 집단 간 뚜렷한 유전적 분화가 존재함을 의미하며, 앞선 FST 분석에서 확인된 집단 간 유전적 차이와도 일관된 결과이다.
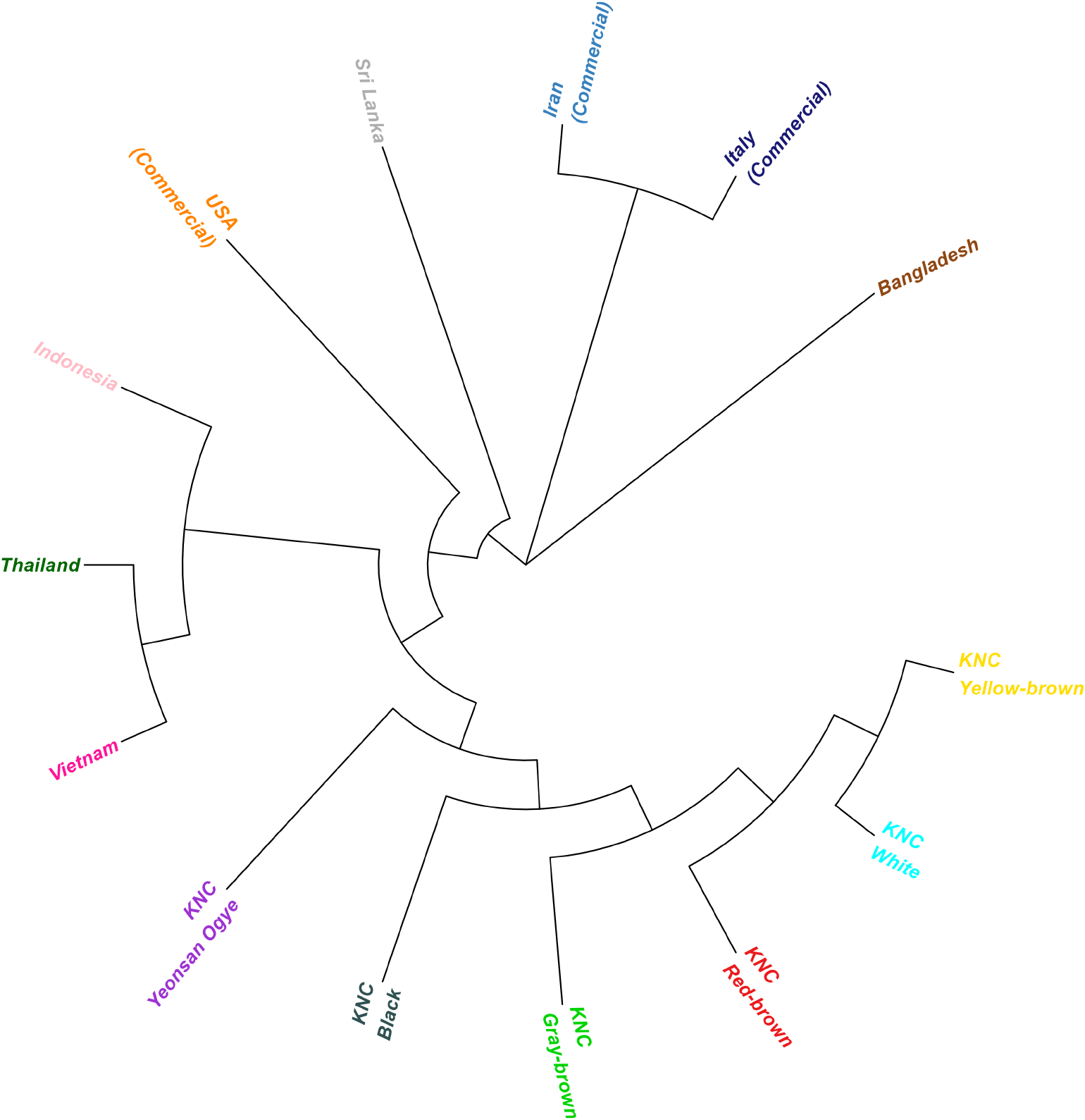
이러한 분리 양상은 집단별 상이한 계통적 기원과 육종 역사를 반영하는 것으로 해석된다. KNC는 1990년대에 복원된 이후 장기간 보존 중심으로 유지되며 선발·개량이 제한적으로 이루어져온 집단이다(Cho et al., 2021). 반면, 실용계 집단은 수십 년간 성장률과 사료효율을 중심으로 강한 인위적 선발이 지속되어 왔으며(Zuidhof et al., 2014), 이러한 개량 과정에서 열내성과 연관된 유전자군도 함께 변화했을 가능성이 크다. 또한 열대 지역 재래닭들은 장기간 고온다습한 환경에서 적응해 온 계통으로(Guo et al., 2022; Rachman et al., 2024), 환경적 선발 압력의 방향이 KNC와 큰 차이가 있다. 이러한 KNC와 외래 집단의 분리 패턴은 단순한 유전적 차이뿐 아니라, 자연적·인위적 선발 압력의 차이, 육종 체계의 차별성, 환경 적응 역사가 복합적으로 작용해 열내성 연관 유전자 구간에서도 집단 간 유전적 분리를 유도했음을 시사한다.
더 나아가, 열내성 반응은 세포 내 신호전달 및 대사 조절뿐만 아니라 면역 및 항원제시 체계에도 영향을 미치는 것으로 보고되어 있다(Monson et al., 2018). 특히 닭의 주조직적합복합체(major histocompatibility complex, MHC)는 질병 저항성과 밀접하게 연관된 대표적인 면역 유전자군으로, 환경 스트레스에 대한 반응에도 중요한 역할을 수행하는 것으로 알려져 있다(Silva and Gallardo, 2020). 이러한 점을 고려하면, 열내성 형질의 생물학적 배경을 해석할 때 대사 및 세포 스트레스 경로뿐 아니라 면역 기반 반응 역시 잠재적인 고려 요소가 될 수 있다. 본 연구에서는 열내성 후보 유전자 영역에 한정된 분석 결과를 제시하였으나, 향후 연구에서 MHC를 포함한 면역 관련 유전자군과 본 연구에서 확인된 유전 변이를 병행하여 평가한다면, 고온 환경에서의 열내성 반응을 보다 다각적으로 이해하는 데 기여할 것으로 기대된다.
적 요
본 연구는 KNC와 외래 집단을 대상으로 열내성과 연관된 19개 유전자 영역 내 존재하는 SNP 변이를 분석하여 집단 간 유전적 구조를 규명하였다. 분석 결과, KNC는 외래 집단과 명확하게 분리되는 구조를 보였으며, 이는 PCA, FST 및 계통수 분석에서 일관되게 확인되었다. 이러한 결과는 KNC가 외래 집단과 구별되는 열내성 관련 유전적 특성을 보유하고 있음을 시사한다. 또한 열내성이 면역 및 항원제시 기전과도 밀접하게 연관된다는 점을 고려하면, 향후 MHC를 포함한 면역 관련 유전자군과의 통합 분석을 통해 KNC의 열내성 메커니즘을 보다 다각적으로 이해할 수 있을 것으로 판단된다. 다만 본 연구는 SNP 기반 후보 유전자 분석으로 표현형 자료가 부재하여, 관찰된 분리 패턴이 실제 열내성 반응에 따른 것인지 혹은 품종 고유의 유전적 구조에 기인한 것인지는 명확히 규명하기에는 제한이 있다. 또한, 전장 유전체 분석이 아닌 후보 유전자 중심의 분석을 수행하였기에, 분석 범위가 제한됨에 따른 선택 편향 가능성을 완전히 배제하기는 어렵다. 그럼에도 본 연구는 KNC의 열내성 유전체 특성을 규명하는 기초자료로서 의의를 가지며, 향후 열내성 개량과 분자마커 기반 선발 전략 수립에 활용될 수 있을 것으로 기대된다.








